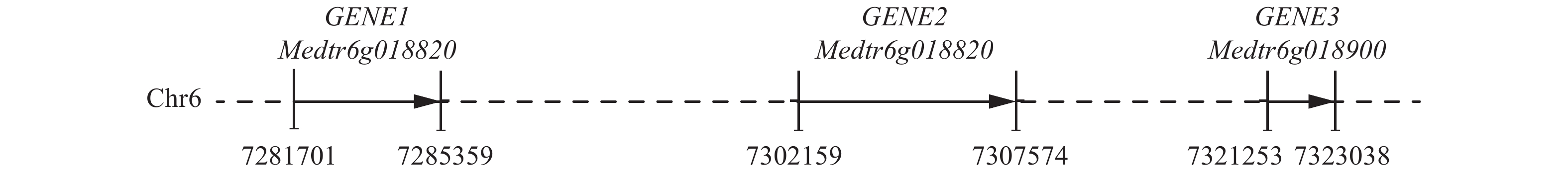
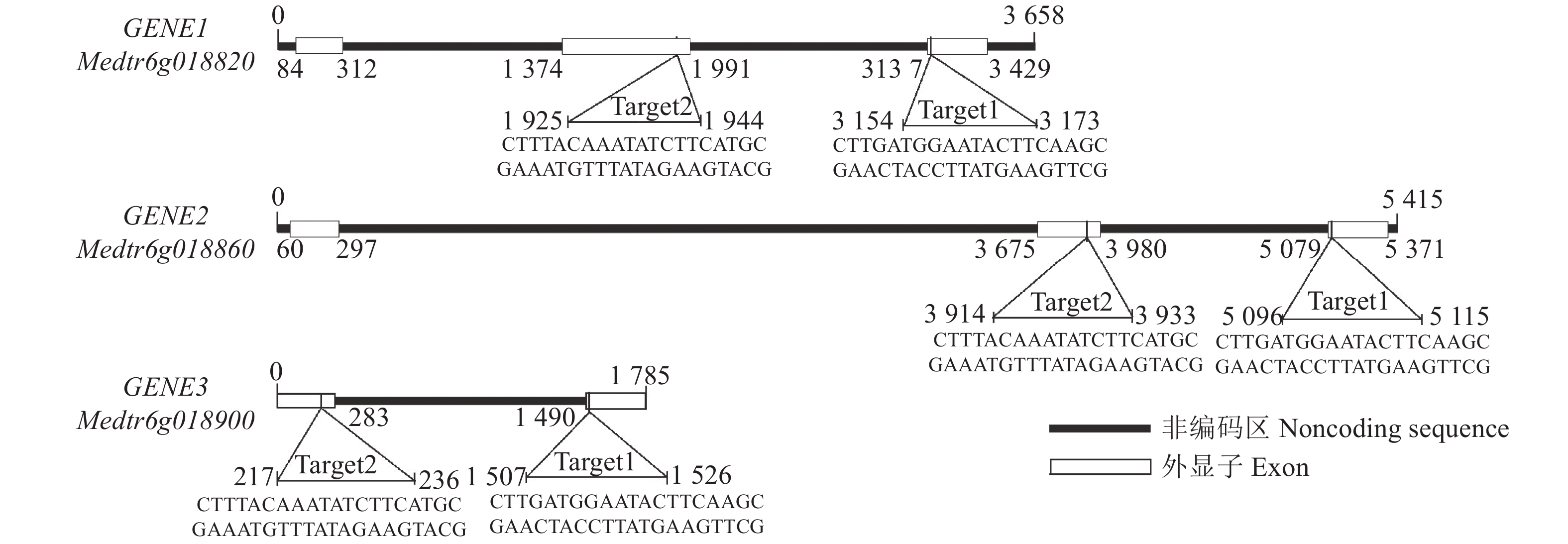
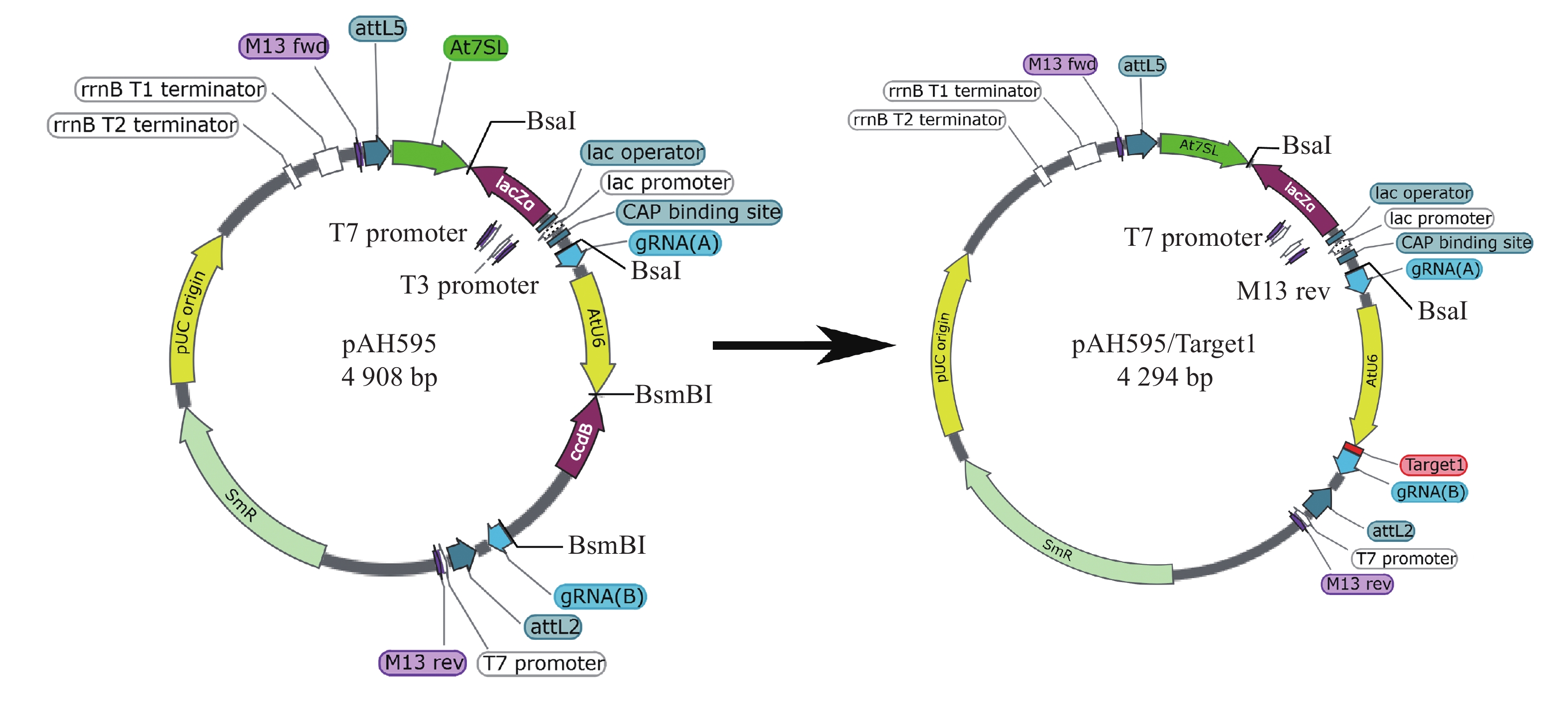
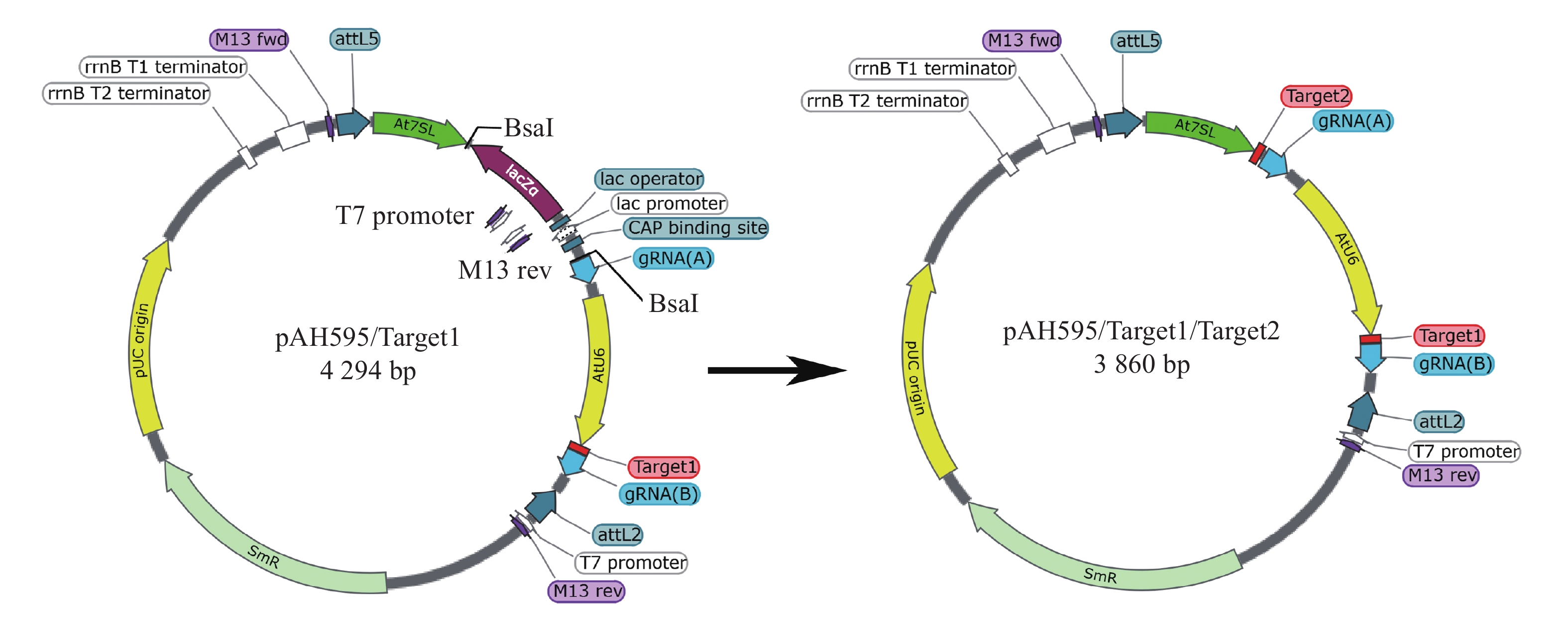
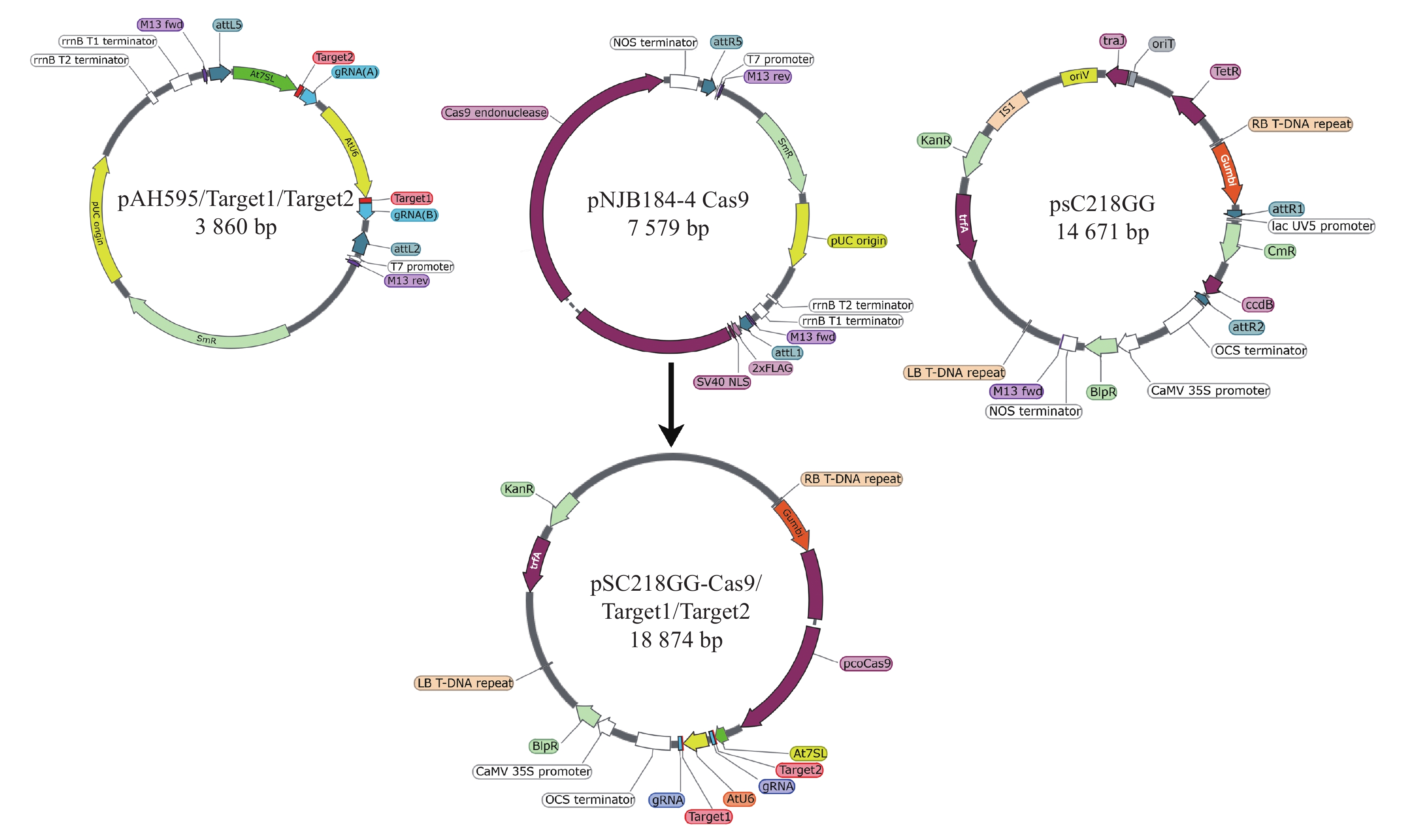
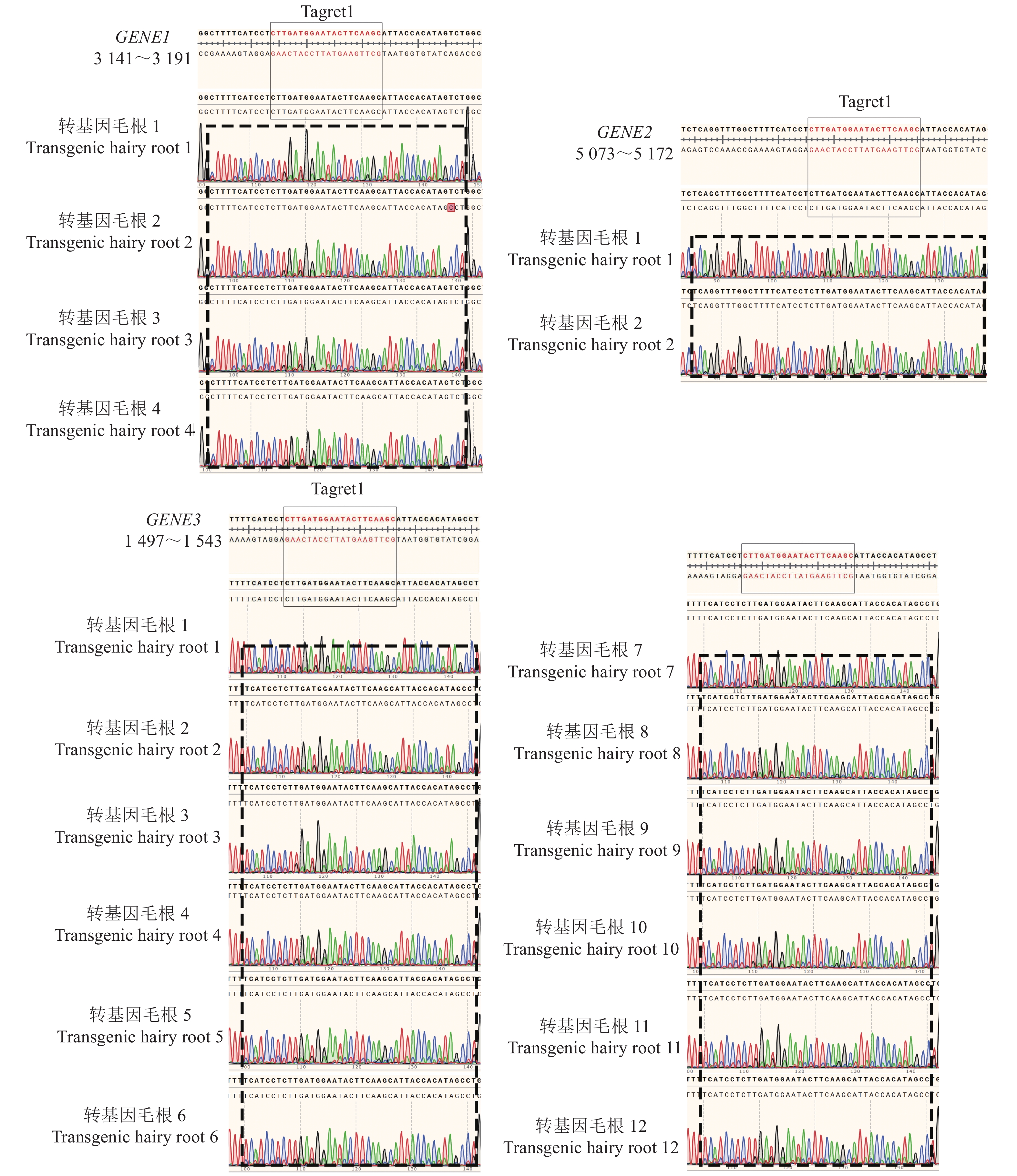
利用CRISPR-Cas9编辑蒺藜苜蓿串联重复基因
English
-
随着我国农业供给侧结构性改革和“粮改饲”政策的实施,大力推广青贮玉米的生产利用,降低动物饲喂成本,提高生产性能,减轻秸秆处理的环境压力,已成为形成种养结合生态循环生产模式的有效途径[1]。青贮玉米(Zea mays)是发展节粮型畜牧业的有效手段,具有生物量高、适口性好、养分消化率高和贮藏时间长等特点[2],是公认的优质粗饲料。青贮前的鲜料中含有一些有害好氧菌,随着氧气消耗,有害菌减少。青贮玉米中加入青贮发酵促进剂,可使乳酸菌迅速成为优势菌群,形成酸性环境并抑制好氧微生物的生长繁殖[3];保证青贮初期发酵所需的乳酸菌数量,使pH迅速下降,蛋白水解受到抑制,青贮料中氨态氮、乙酸(acetic acid, AA)和丁酸(butyric acid, BA)浓度降低,乳酸(lactic acid, LA)浓度增加[4]。研究表明,去穗玉米秸秆青贮中添加发酵促进剂宜生贮宝(Sila-Max)和宜生贮康(Sila-Mix)均可提高发酵产物的干物质(dry matter, DM)含量[5];代寒凌等[6]研究指出,Sila-Max和Sila-Mix处理组的LA含量显著高于未添加对照组,pH和氨态氮(NH3-N)显著低于对照组,青贮营养品质和发酵品质均优于对照组。青贮玉米时添加Sila-Max可提高粗蛋白(crude protein, CP)、总可消化养分(total digestible nutrients, TDN)、DM含量,降低中性洗涤纤维(neutral detergent fiber, NDF)含量,有效提高青贮饲料品质[7]。
玉米植株的营养成分含量在其成熟过程中不断变化,适宜收获期对青贮营养品质及其发酵品质起决定性作用[8]。收获期过晚使纤维消化率降低,造成青贮营养价值降低[9]。选择适宜的收获期对青贮营养品质把控具有重要的作用,有研究表明,全株玉米随着收获期延迟CP、AA和LA含量逐渐递减,粗脂肪(ether extract, EE)和水溶性碳水化合物(water soluble carbohydrates, WSC)含量先上升后下降[10]。在多花黑麦草(Lolium multiflorum)研究中发现,随着收获期的延迟,DM、酸性洗涤纤维(acid detergent fiber, ADF)、NDF含量呈增长趋势,CP含量和48 h干物质体外消化率(48 h in vitro dry matter digestibility, IVDMD48 h)呈下降趋势,WSC含量呈先上升后下降[11]。在全株玉米中,随着收获期的延迟,DM、淀粉(starch, ST)含量显著增加[12]。目前已有大量关于Sila-Max和Sila-Mix分别对全株玉米营养品质影响的研究,但有关Sila-Max和Sila-Mix作用效果对比的研究很少。为此,本研究对添加Sila-Max和Sila-Mix及不同收获期下全株玉米营养品质进行综合比较,以期筛选出最优青贮发酵促进剂和收获期,为进一步提高玉米青贮饲料营养品质和适宜收获期提供理论依据。
1. 材料和方法
1.1 试验地概况
试验地位于甘肃省庆阳市环县木钵镇胡家湾村(36°22′ N,107°27′ E),海拔1 180 m,年平均气温9.2 ℃,年平均降水量300 mm左右,属温带大陆性半干旱气候。0 − 10 cm土层土壤有机质含量为11.10 g·kg−1,全氮含量为0.81 g·kg−1,速效磷含量为7.30 mg·kg−1,速效钾含量为194.00 mg·kg−1,pH 8.1。
1.2 试验材料
1.2.1 青贮原料
选用‘豫青贮23’专用青贮玉米品种为试验材料,该品种由河南省大京九种业有限公司选育而成,适宜在北京、天津武清、河北北部(张家口除外)、辽宁东部、吉林中南部和黑龙江第一积温带春播区种植。试验用玉米于2018年5月1日种植,田间管理同当地大田,分别在1/2、2/3、3/4乳线期收割,然后称取500 g粉碎后的全株玉米装入30 cm × 20 cm的聚乙烯青贮袋中进行青贮。
1.2.2 青贮发酵促进剂
所用青贮发酵促进剂宜生贮宝(贮宝)、宜生贮康(贮康)由美国农业部与甘肃农业大学共同研发。其中贮宝含有植物乳酸菌、丙酸杆菌、纯化纤维素酶,乳酸菌 ≥ 2 × 1011 cfu·g−1;贮康含有乳酸菌、碳酸钙、黑曲霉,乳酸菌 ≥ 1.8 × 106 cfu·g−1。此外,贮宝和贮康含有同型发酵乳酸菌、异型发酵乳酸菌和枯草芽孢杆菌(α-淀粉酶)。
1.3 试验方法
试验采用收获期和促进剂双因子交互设计,对不同收获期(1/2、2/3、3/4乳线期)全株青贮玉米进行两种促进剂(贮宝和贮康)处理下的青贮试验,不添加任何青贮发酵促进剂作为对照组(CK)。试验每个收获期分为3个处理,3个收获期共9个处理,每个处理3次重复。
不同收获期的全株玉米粉碎后,两种促进剂的添加量按照产品说明添加(贮宝粉末按2.5 mg·kg−1均匀加入,贮康颗粒按500 mg·kg−1均匀加入)。均匀喷洒添加剂后称取500 g粉碎后的全株玉米装入同样的聚乙烯青贮袋中,用真空包装机抽真空后密封,在暗处恒温(20 ℃)贮藏60 d后测定各指标。
1.4 测定指标与方法
1.4.1 感官评定
根据德国农业协会(DLG)青贮感官评分标准[13]对青贮进行感官鉴定,对青贮的气味(14分)、质地(4分)、色泽(2分)进行评分,根据总分把青贮分为Ⅰ级(优良,16~20分)、Ⅱ级(尚好,10~15分)、Ⅲ级(中等,5~9分)及Ⅳ级(腐败,0~4分) 4个等级。
1.4.2 营养指标的测定
采用张丽英[14]的方法测定DM、CP、EE及Ash含量;按照范氏(Van Soest)洗涤纤维法[15]测定NDF和ADF含量;WSC含量测定按照蒽酮−硫酸比色法[16]测定;ST含量采用AOAC[17]的方法测定;采用雷磁PHS-3C精密pH计测定pH;氨态氮含量采用苯酚次氧酸钠比色法[18]测定,LA、AA及丁酸(butyric acid,BA)含量使用安捷伦 (Agilent1100) 液相色谱仪测定[19]。
$\begin{split}& \mathrm{D}\mathrm{M}\mathrm{回}\mathrm{收}\mathrm{率}= \\& 1-\dfrac{\mathrm{青}\mathrm{贮}\mathrm{前}\mathrm{袋}\mathrm{中}\mathrm{样}\mathrm{品}\mathrm{D}\mathrm{M}\mathrm{量}-\mathrm{青}\mathrm{贮}\mathrm{后}\mathrm{袋}\mathrm{中}\mathrm{样}\mathrm{品}\mathrm{D}\mathrm{M}\mathrm{量}}{\mathrm{青}\mathrm{贮}\mathrm{前}\mathrm{袋}\mathrm{中}\mathrm{样}\mathrm{品}\mathrm{D}\mathrm{M}\mathrm{量}}\times 100\mathrm{\text{%}}\mathrm{。} \end{split}$


IVDMD48 h和48 h中性洗涤纤维消化率(48 h in vitro neutral detergent fiber digestibility, IVNDFD48 h)采用活体外人工瘤胃法测定[20]。
$\begin{split} &{\text{干物质体外消化率}}({\rm{IVDMD}})=\\& \dfrac{({\text{消化前样品}}{\rm{DM}}{\text{重}}-{\text{消化后样品}}{\rm{DM}}{\text{重}})}{{\text{消化前样品}}{\rm{DM}}{\text{重}}} \times 100\mathrm{\text{%}}\text{;} \end{split}$


$\begin{split} &{\text{中性洗涤纤维消化率}}({\rm{IVNDFD}})=\\& \dfrac{({\text{消化前样品}}{\rm{NDF}}{\text{重}}-{\text{消化后样品}}{\rm{NDF}}{\text{重}})}{{\text{消化前样品}}{\rm{NDF}}{\text{重}}}\times 100\mathrm{\text{%}} ;\end{split} $


$ {\rm{TDN}} = {\rm{DCP}} + {\rm{DCF}} + 2.25{\rm{DEE}} + {\rm{DNFE}}。 $


式中:TDN为总可消化养分,DCP (digestible crude protein)为可消化粗蛋白,DCF (digestible compensating fiber)为可消化粗纤维,DEE (digestible ether extract)为可消化粗脂肪及DNFE (digestible nitrogen free extract)为可消化无氮浸出物的百分含量[21]。
1.5 数据统计分析
采用Excel 2016软件对数据进行初步处理,用SPSS 24.0完成数据分析,数据均以“平均值 ± 标准误”表示,对促进剂和收获期进行双因素方差分析 (Two-way ANOVA),对同一促进剂不同收获期处理、同一收获期不同促进剂处理进行单因素方差分析(One-way ANOVA),并用 Duncan 法对各项指标进行多重比较 (P < 0.05)。采用灰色关联度法[22]对青贮玉米营养品质进行综合分析。
2. 结果分析
2.1 青贮原料的化学成分
随着收获期延迟,全株青贮玉米3/4乳线期的DM、EE、Ash、NDF、ADF、ST显著高于1/2乳线期(P < 0.05),1/2乳线期的CP、IVDMD48 h、IVNDFD48 h、TDN显著高于3/4乳线期(P < 0.05),2/3乳线期的WSC显著高于其他两个时期(P < 0.05)(表1)。
表 1 不同收获期青贮原料的化学成分Table 1. Chemical composition of silage materials in different harvest stages指标
Parameter收获期 Haverst stage 1/2乳线期
1/2 milk line period2/3乳线期
2/3 milk line period3/4乳线期
3/4 milk line period干物质 Dry matter (DM)/% 27.77 ± 0.26c 30.43 ± 0.12b 31.93 ± 0.13a 粗蛋白 Crude protein (CP)/% 8.25 ± 0.12a 6.45 ± 0.47b 5.51 ± 0.17c 粗脂肪 Ether extract (EE)/% 4.44 ± 0.36b 7.29 ± 0.25b 8.97 ± 0.72a 粗灰分 Ash/% 6.19 ± 0.37b 8.71 ± 0.46b 15.29 ± 1.21a 中性洗涤纤维 Neutral detergent fiber (NDF)/% 44.13 ± 0.17b 44.80 ± 0.57b 53.09 ± 0.97a 酸性洗涤纤维 Acid detergent fiber (ADF)/% 26.68 ± 0.11b 30.19 ± 0.90b 42.69 ± 2.00a 淀粉 Starch (ST)/% 7.35 ± 1.42c 18.95 ± 1.04b 25.48 ± 0.15a 水溶性碳水化合物 Water soluble carbohydrate (WSC)/% 6.47 ± 0.26b 9.51 ± 0.58a 8.29 ± 0.54b 48 h干物质体外消化率
48 h in vitro dry matter digestibility (IVDMD48 h)/%90.33 ± 0.33a 89.33 ± 0.88a 84.67 ± 0.33b 48 h中性洗涤纤维消化率
48 h in vitro neutral detergent fiber digestibility (IVNDFD48 h)/%72.33 ± 0.67a 68.00 ± 0.00b 63.67 ± 1.67c 总可消化养分 Total digestible nutrients (TDN)/% 70.00 ± 0.00a 69.67 ± 0.33a 62.67 ± 0.67b 同行不同小写字母表示不同收获期差异显著(P < 0.05)。
Different lowercase letters within the same row indicate significant differences between different harvest stages at the 0.05 level.2.2 青贮发酵促进剂和收获期对全株玉米青贮质量感官评分的影响
各处理组均有较强的芳香味,色泽均为黄绿色,接近全株青贮玉米原料原色,茎叶分明,结构保存良好,在聚乙烯青贮袋中未发现成块现象,综合评价等级均为Ⅰ级(表2)。
表 2 不同处理下玉米青贮感官评价得分Table 2. Sensory evaluation score of corn silage under different treatments收获期
Harvest stage项目
Item对照
Control宜生贮宝
Sila-Max宜生贮康
Sila-Mix1/2乳线期
1/2 milk line period气味 Odor 13 14 14 质地 Texture 3 3 3 色泽 Color 1 1 1 总分 Total score 17 18 18 2/3乳线期
2/3 milk line period气味 Odor 13 14 14 质地 Texture 3 3 3 色泽 Color 1 2 1 总分 Total score 17 19 18 3/4乳线期
3/4 milk line period气味 Odor 13 14 14 质地 Texture 3 3 3 色泽 Color 1 1 2 总分 Total score 17 18 19 2.3 青贮发酵促进剂和收获期对全株青贮玉米发酵品质的影响
方差分析(表3)表明,青贮发酵促进剂和收获期的交互作用对所有指标均无显著影响(P > 0.05)。收获期对LA、AA、BA及DMR的影响极显著(P < 0.01),对pH影响显著(P < 0.05),而对氨态氮含量的影响不显著(P > 0.05)。青贮发酵促进剂对氨态氮、LA、AA及DMR的影响极显著(P < 0.01),而对pH、BA含量的影响不显著(P > 0.05)。
表 3 不同处理对全株青贮玉米发酵品质的影响Table 3. Effects of different treatments on fermentation quality of whole silage maize收获期
Haverst stage处理
TreatmentpH 氨态氮
NH3-N/%乳酸
Lactic
acid (LA)/%乙酸
Acetic
acid (AA)/%丁酸
Butyric
acid (BA)/%干物质回收率
Dry matter
recovery (DMR)/%1/2乳线期
1/2 milk line
period对照 Control 3.81 ± 0.52 0.39 ± 0.27A 4.91 ± 0.72B 3.06 ± 0.48Aa 0.04 ± 0.03A 90.97 ± 0.00Bb 宜生贮宝 Sila-Max 3.78 ± 0.52 0.37 ± 0.21Aa 5.50 ± 0.28A 3.05 ± 0.88Aa 0.04 ± 0.00B 97.46 ± 0.01Aa 宜生贮康 Sila-Mix 3.81 ± 0.06 0.21 ± 0.02Bb 5.63 ± 0.37Aa 2.30 ± 0.21Ba 0.04 ± 0.00AB 92.46 ± 0.00Bb 2/3乳线期
2/3 milk line
period对照 Control 3.86 ± 0.01 0.41 ± 0.06A 4.52 ± 0.15B 2.57 ± 0.12Ab 0.05 ± 0.00 91.00 ± 0.01b 宜生贮宝 Sila-Max 3.83 ± 0.01 0.37 ± 0.36Aa 5.38 ± 0.11A 2.16 ± 0.77Bb 0.04 ± 0.00 93.35 ± 0.01b 宜生贮康 Sila-Mix 3.85 ± 0.70 0.23 ± 0.04Bb 5.27 ± 0.09Aab 1.89 ± 0.05Ca 0.04 ± 0.00 91.44 ± 0.01b 3/4乳线期
3/4 milk line
period对照 Control 3.94 ± 0.20A 0.36 ± 0.26 4.35 ± 0.22B 1.50 ± 0.19c 0.05 ± 0.00 95.90 ± 0.01a 宜生贮宝 Sila-Max 3.85 ± 0.01B 0.22 ± 0.87b 5.17 ± 0.24A 1.36 ± 0.32c 0.05 ± 0.01 96.70 ± 0.00a 宜生贮康 Sila-Mix 3.86 ± 0.02B 0.31 ± 0.03a 4.56 ± 0.24Bb 1.08 ± 0.05b 0.05 ± 0.01 96.30 ± 0.01a 收获期 Harvest stages (H) * NS ** ** ** ** 促进剂 Promoter (P) NS ** ** ** NS ** P × H NS NS NS NS NS NS 同列不同大写字母表示相同收获期不同促进剂之间差异显著(P < 0.05),同列不同小写字母表示相同促进剂不同收获期差异显著(P < 0.05),无字母表示无显著差异,*表示差异显著(P < 0.05),**表示差异极显著(P < 0.01),NS表示差异不显著(P > 0.05);下表同。
Different capital letters within the same column for the same harvest stages indicate significant difference between different promoters at the 0.05 level, different lowercase letters within the same column for the same promoter indicate significant difference between different harvest stages at the 0.05 level, No letters means no significant difference, *, P < 0.05; * * , P < 0.01; NS, P > 0.05; this is applicable for the following tables as well.相同收获期不同促进剂间,3/4乳线期CK组pH显著高于其他组(P < 0.05)。1/2、2/3乳线期贮康组氨态氮含量显著低于其他组。1/2、2/3乳线期的CK组LA含量显著低于其他组,3/4乳线期贮宝组LA含量显著高于其他组。1/2乳线期贮康组AA含量显著低于其他组,2/3乳线期CK组AA含量显著高于其他组。1/2乳线期CK组BA含量显著高于贮宝组,贮宝组DMR显著高于其他组。
相同促进剂不同收获期之间,对于CK组,1/2乳线期AA含量显著高于其他时期(P < 0.05),3/4乳线期DMR显著高于其他时期。对于贮宝组,3/4乳线期氨态氮含量显著低于其他时期,1/2乳线期AA含量显著高于其他时期,2/3乳线期DMR显著低于其他时期。对于贮康组,3/4乳线期氨态氮含量显著高于其他时期,3/4乳线期LA含量显著低于1/2乳线期,3/4乳线期AA含量显著低于其他时期,3/4乳线期DMR显著高于其他时期。
2.4 青贮发酵促进剂和收获期对全株玉米青贮的营养成分含量的影响
方差分析(表4)表明,收获期对DM、CP、EE、Ash、NDF、ADF、ST、WSC含量影响极显著(P < 0.01),青贮发酵促进剂对DM、CP、WSC含量影响极显著(P < 0.01),对ST含量影响显著(P < 0.05),对其他营养指标均无显著影响(P > 0.05)。
表 4 不同处理对全株青贮玉米营养成分的影响Table 4. Effects of different treatments on nutrient components of whole silage maize收获期
Haverst
stage处理
Treatment干物质
Dry
matter (DM)/%粗蛋白
Crude protein (CP)/%粗脂肪
Ether
extract (EE)/%粗灰分
Ash/%中性洗涤纤维
Neutral detergent fiber (NDF)/%酸性洗涤纤维
Acid detergent fiber (ADF)/%淀粉
Starch (ST)/%水溶性碳水化合物
Water soluble carbohydrate (WSC)/%1/2乳线期
1/2 milk line
period对照 Control 25.26 ± 0.25Bc 8.20 ± 0.03Ba 2.31 ± 0.11c 5.48 ± 0.23c 38.10 ± 0.90c 24.50 ± 0.35c 14.27 ± 0.70c 3.02 ± 0.15b 宜生贮宝 Sila-Max 27.06 ± 0.14Ac 9.25 ± 0.07Aa 2.57 ± 0.12b 5.44 ± 0.04c 36.88 ± 0.36b 23.87 ± 0.60c 15.65 ± 0.60c 3.36 ± 0.24b 宜生贮康 Sila-Mix 25.68 ± 0.36Bc 9.13 ± 0.07Aa 2.34 ± 0.20b 5.56 ± 0.33b 37.38 ± 0.54c 24.21 ± 0.31c 15.89 ± 1.16c 3.34 ± 0.05b 2/3乳线期
2/3 milk line
period对照 Control 27.69 ± 0.10b 8.30 ± 0.09a 3.20 ± 0.11Bb 7.86 ± 0.12Bb 42.93 ± 1.47b 32.56 ± 0.11b 18.60 ± 0.24b 4.02 ± 0.18a 宜生贮宝 Sila-Max 28.41 ± 0.34b 8.53 ± 0.04b 3.21 ± 0.07Bb 7.90 ± 0.20Bb 42.46 ± 1.73a 30.40 ± 0.88b 22.33 ± 1.73b 4.29 ± 0.06a 宜生贮康 Sila-Mix 27.82 ± 0.18b 8.41 ± 0.08b 4.90 ± 0.30Aa 8.89 ± 0.23Aa 42.51 ± 0.40b 31.05 ± 0.94b 22.51 ± 1.92b 4.68 ± 0.62a 3/4乳线期
3/4 milk line
period对照Control 30.61 ± 0.02a 5.36 ± 0.63b 5.26 ± 0.31a 9.54 ± 0.21a 47.55 ± 0.50Aa 37.11 ± 1.05a 27.53 ± 1.07a 1.40 ± 0.07Bc 宜生贮宝 Sila-Max 30.87 ± 0.26a 6.13 ± 0.26c 5.65 ± 0.37a 9.54 ± 0.21a 46.09 ± 0.60Ba 35.85 ± 0.33a 28.70 ± 1.05a 2.56 ± 0.17Ac 宜生贮康 Sila-Mix 30.74 ± 0.24a 6.13 ± 0.43c 5.63 ± 0.73a 10.01 ± 0.84a 46.36 ± 0.50Ba 36.24 ± 0.95a 29.69 ± 0.07a 2.66 ± 0.17Ac 收获期 Harvest stages (H) ** ** ** ** ** ** ** ** 促进剂 Promoter (P) ** ** NS NS NS NS * ** P × H * NS ** NS NS NS NS NS 相同收获期不同促进剂间,1/2乳线期贮宝组DM含量显著高于其他组(P < 0.05),CK组CP含量显著低于其他组。2/3乳线期贮康组EE含量显著高于其他组,贮康组Ash含量显著高于其他组。3/4乳线期贮宝和贮康组NDF含量显著低于CK组,贮宝和贮康组DMR显著高于CK组。
相同促进剂不同收获期间,对于CK组,3/4乳线期DM、EE、Ash、ADF及ST含量显著高于其他时期(P < 0.05),CP含量显著低于其他时期,1/2乳线期NDF含量显著低于其他时期,2/3乳线期WSC含量显著高于其他时期。对于贮宝组,3/4乳线期DM、EE、Ash、NDF、ADF及ST含量显著高于其他收获期,CP含量显著低于其他时期,2/3乳线期WSC含量显著高于其他时期。对于贮康组,3/4乳线期DM、EE、Ash、NDF、ADF及ST含量显著高于其他时期,CP含量显著低于其他时期,2/3乳线期WSC含量显著高于其他收获期。
2.5 青贮发酵促进剂和收获期对全株青贮玉米消化率及总可消化养分的影响
方差分析(表5)表明,青贮发酵促进剂和收获期的交互作用对消化率及总可消化养分不显著(P > 0.05),收获期对IVDMD48 h、IVNDFD48 h、TDN影响极显著(P < 0.01),青贮发酵促进剂对IVDMD48 h、IVNDFD48 h及 TDN影响不显著(P > 0.05)。
表 5 不同处理对全株青贮玉米消化率和总可消化养分的影响Table 5. Effects of different treatments on the digestibility and total digestible nutrients of whole silage maize收获期
Harvest stage处理
Treatment48 h干物质体外消化率 (IVDMD48 h)
48 h in vitro dry matter digestibility/%48 h中性洗涤纤维消化率 (IVNDFD48 h)
48 h in vitro neutral detergent fiber digestibility/%总可消化养分 (TDN)
Total digestible nutrients /%1/2乳线期
1/2 milk line period对照 Control 88.33 ± 0.67Ba 65.67 ± 0.33a 71.00 ± 0.58a 宜生贮宝 Sila-Max 89.67 ± 0.33Aa 66.00 ± 0.33a 71.67 ± 0.33a 宜生贮康 Sila-Mix 88.67 ± 0.33Ba 66.33 ± 0.33 71.67 ± 0.33a 2/3乳线期
2/3 milk line period对照组 Control 86.00 ± 1.52ab 65.33 ± 0.33a 67.67 ± 0.33b 宜生贮宝 Sila-Max 86.33 ± 1.20b 65.33 ± 0.67a 68.33 ± 0.88b 宜生贮康 Sila-Mix 86.67 ± 1.37b 65.67 ± 1.03 68.67 ± 1.37b 3/4乳线期
3/4 milk line period对照 Control 84.00 ± 0.58b 64.00 ± 0.00Bb 65.00 ± 0.58c 宜生贮宝 Sila-Max 84.33 ± 0.33b 64.00 ± 0.00Bb 65.33 ± 0.33c 宜生贮康 Sila-Mix 84.33 ± 0.33c 65.00 ± 0.58A 65.67 ± 0.33c 收获期 Harvest stages (H) ** ** ** 促进剂 Promoter (P) NS NS NS P × H NS NS NS 相同收获时期不同促进剂间,1/2乳线期贮宝组IVDMD48 h显著高于其他组(P < 0.05),3/4乳线期贮康组IVNDFD48 h显著高于其他组。
相同促进剂不同收获期间,对于CK组,1/2乳线期IVDMD48 h显著高于3/4乳线期(P < 0.05),TDN显著高于其他时期,3/4乳线期IVNDFD48 h显著低于其他时期。对于贮宝组,1/2乳线期IVDMD48 h、TDN显著高于其他时期,3/4乳线期IVNDFD48 h显著低于其他收获期。对于贮康组,1/2乳线期IVDMD48 h、TDN显著高于其他时期。
2.6 各营养品质的灰色关联度分析
根据加权值筛选出的适宜收获期和青贮发酵促进剂为2/3乳线期添加贮康(表6)。
表 6 各营养品质灰色关联度分析Table 6. Gray correlation analysis of each nutritional quality收获期
Haverst stage处理
Treatment加权关联值
Weighted
correlation排序
Sort1/2乳线期
1/2 milk line
period对照 Control 0.041 19 Ⅷ 宜生贮宝 Sila-Max 0.050 66 Ⅴ 宜生贮康 Sila-Mix 0.052 49 Ⅲ 2/3乳线期
2/3 milk line
period对照 Control 0.040 56 Ⅸ 宜生贮宝 Sila-Max 0.047 81 Ⅵ 宜生贮康 Sila-Mix 0.056 57 Ⅰ 3/4乳线期
3/4 milk line
period对照 Control 0.042 38 Ⅶ 宜生贮宝 Sila-Max 0.051 55 Ⅳ 宜生贮康 Sila-Mix 0.055 50 Ⅱ 3. 讨论与分析
3.1 青贮发酵促进剂对全株青贮玉米感官鉴定的影响
对青贮发酵产物进行直接的感官评价,可快速的从质地、色泽、气味方面判断青贮品质的优劣,从而判断青贮是否成功。张相伦等[23]研究乳酸菌制剂对全株玉米的感官鉴定发现,试验各组青贮感官评价综合评分均在16~20分,达到优级标准,青贮效果均较好。袁仕改等[24]研究不同添加剂对青贮玉米的感官鉴定发现,所有试验组感官评价均为Ⅰ级优良。本研究中添加青贮发酵促进剂与未添加组全株青贮玉米感官评价均为Ⅰ级优良,且贮宝和贮康组均高于CK组。这表明全株玉米青贮时,不使用添加剂也能得到感官较好的青贮饲料,与上述结果一致。
3.2 青贮发酵促进剂和收获期对全株青贮玉米发酵品质的影响
pH、氨态氮和乳酸含量是衡量饲料发酵品质的重要指标。优质青贮饲料具有较高的LA含量和较低pH、BA含量[25],高LA含量和低pH能够抑制有害菌生长,降低蛋白质降解[26]。通常优质青贮饲料的 pH 在3.8~4.2 [27]。本研究中各处理组pH都低于4.0,达到了优质青贮饲料的要求,而且试验组pH显著低于CK组,贮康组高于贮宝组,这是因为贮康除了含有贮宝所含有的菌类之外还含有25.0%~29.5%的CaCO3,CaCO3可以通过提高青贮发酵产物的有机酸含量及pH,提高饲料的适口性,中和酸环境中的一些氢离子[4]。氨态氮是反映青贮过程中蛋白质降解程度的指标,其含量越高表明蛋白氮降解越多,青贮发酵品质越差。张适等[28]研究指出,添加促进剂的各处理组中氨态氮含量均显著降低,蛋白分解较少。本研究中贮宝和贮康组的氨态氮含量显著低于CK组,与张适等[28]研究结果一致。有研究发现,添加青贮发酵促进剂可显著降低青贮pH,提高LA含量[4, 29]。本研究中,试验组LA含量显著高于CK组,表明青贮发酵促进剂可以使青贮玉米产生大量LA。王旭哲等[30]发现,青贮中加入同型发酵乳酸菌,LA含量较高,所产生的AA含量较少,本研究中试验组AA含量显著低于CK组,原因是青贮发酵促进剂贮宝和贮康属于同型乳酸菌,在发酵过程中抑制了AA的产生,此结果与王旭哲等[30]报道一致。刘辉等[31]研究表明,试验中添加青贮发酵促进剂在两种凋萎青贮中获得了积极的效果,DMR均有不同程度提高,本研究试验组DMR显著高于CK组, 表明两种青贮发酵促进剂有助于提高全株青贮玉米的发酵品质。
pH与青贮原料中的WSC含量有关,通常来说较高的WSC为乳酸菌发酵提供了大量营养物质, 促进乳酸菌活动, 进而产生大量的乳酸来降低pH[32]。本研究中随着收获期延迟WSC含量降低,pH在CK组显著升高,试验组有相对升高的趋势,这表明收获期延迟会使全株青贮玉米中pH升高。有研究表明青贮中LA含量随着收获期延迟降低[7]。本研究贮康组3/4乳线期LA含量显著低于1/2乳线期,CK和贮宝组也逐渐降低,其原因是在收获早期,青贮中水分含量多,微生物活动频繁,但随着收获期延迟水分含量下降,微生物活动减弱,进而会使全株青贮玉米中乳酸含量降低。收获期延迟减少了全株青贮玉米中蛋白质降解,从而降低了氨态氮含量[33],本研究也得出相同结论。因为其中氨主要由梭状芽胞杆菌产生[34],青贮玉米中水分含量的降低能够抑制梭状芽胞杆菌的生长,进而会减缓蛋白质的降解与氨的产生[1]。焉石[35]报道指出全株青贮玉米在蜡熟期末期BA含量显著升高,本研究中3/4乳线期BA含量也有升高的趋势,原因是贮宝和贮康中含有相同的乳酸菌,收获期延迟导致LA含量降低且pH升高,产生大量BA,青贮品质变差。
3.3 青贮发酵促进剂和收获期对全株青贮玉米营养品质的影响
全株青贮玉米的营养品质是评价青贮好坏的一项重要指标。青贮时添加发酵促进剂可以提高CP含量[28]。研究表明,在全株青贮玉米中添加青贮发酵促进剂可显著提高DM、CP、ST含量[25]。本研究试验组DM、CP、ST含量显著高于CK组,与前人研究结果一致[24, 28],原因是青贮过程中添加促进剂可快速启动乳酸菌发酵并产生大量LA,快速降低pH,抑制有害菌产生,从而减少DM、CP等营养物质的损耗 [36-37]。ADF通常与动物消化率负相关,其含量越低,饲草消化率越高,饲用价值越大[38]。有研究表明,在全株青贮玉米、大麦中添加不同发酵类型的青贮发酵促进剂,使ADF和NDF含量显著下降[39-40],本研究中贮宝和贮康组的ADF和NDF含量显著低于CK组,其原因是贮宝和贮康中含有纤维素酶,可降解植物细胞壁,与Jones等[39]和Kung等[40]研究结果一致。青贮玉米中含有丰富的WSC,青贮发酵促进剂可抑制有害微生物的产生,促进LA发酵,从而减少了WSC含量的损失,而且青贮发酵促进剂为同质型乳酸菌,乳酸的积累导致部分纤维素和半纤维素通过酸解作用被降解为WSC[4,41],本研究中试验组WSC显著高于CK组,也证实了这一点。受粗饲料中纤维素含量程度的影响,IVDMD48 h越高,粗饲料品质越好。本研究中添加促进剂对IVDMD48 h、IVNDFD48 h及TDN均有促进作用,可以有效降解青贮玉米中的粗纤维,进而提高消化率。
收获期延迟导致青贮玉米的生物量积累,养分浓度都相对增加,纤维化程度增加,消化率降低。朱慧森等[42]指出,从籽粒形成期到完熟期DM、ST含量显著增加。本研究发现3/4乳线期的DM、ST含量显著高于其他两个时期,与其研究结果相一致。植株在衰老过程中叶绿素、维生素等蛋白类物质会降低[43],籽粒形成期玉米青贮饲料CP含量极显著高于完熟期。本研究中3/4乳线期的CP含量显著低于其他时期,收获期延迟CP含量降低。WSC含量与发酵品质显著正相关[44],WSC等有机物的消耗会改变青贮玉米化学成分的比例,可能是导致青贮后化学成分发生变化的主要原因。杨大盛等[45]发现从乳熟中期到蜡熟中期,WSC含量先上升后降低,本研究中WSC含量在2/3乳线期达到最高,在3/4乳线期显著降低,其原因可能与玉米生长过程中营养物质积累和转化有关。陶春卫等[46]指出,TDN与NDF含量显著负相关,随着收获期延迟,纤维化程度增加,IVDMD48 h和IVNDFD48 h降低,本研究中3/4乳线期IVDMD48 h、IVNDFD48 h 及TDN显著低于其他两个时期,其原因是随着生育期的推进,细胞内容物逐渐减少,细胞壁成分增加,这是植物衰老的普遍规律[11]。因此,收获期延迟会导致青贮饲料品质变差,及时在最佳收获期进行收割,可保证青贮营养物质的最大利用。
3.4 各营养成分的灰色关联度分析
灰色关联法可以对系统中的各组要素进行全面的分析,得出参考数列与各比较数列之间的灰色关联度,其比较数列与参考数列的相关性越大,其发展方向和速越接近参考数列,与参考数列的关系越密切,说明整体效果越好[47]。本研究结果显示2/3乳线期贮康综合评价最好。
4. 结论
青贮发酵促进剂对全株青贮玉米的感官质量评价无显著影响,等级均为Ⅰ级。青贮发酵促进剂可提高全株青贮玉米营养品质,收获期延迟会降低营养品质,根据灰色关联度分析得出,2/3乳线期最适宜收获,并且青贮发酵促进剂宜生贮康(Sila-Mix)可在生产实践应用中推广使用。
参考文献
[1] BELHAJ K, CHAPARRO-GARCIA A, KAMOUN S, PATRON N J, NEKRASOV V. Editing plant genomes with CRISPR/Cas9. Current Opinion in Biotechnology, 2015, 32(2): 76-84.
[2] BALTES N J, VOYTAS D F. Enabling plant synthetic biology through genome engineering. Trends in Biotechnology, 2015, 33(2): 120-131. doi: 10.1016/j.tibtech.2014.11.008
[3] JIANG W Y, BIKARD D, COX D, ZHANG F, MARRAFFINI L A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. doi: 10.3724/SP.J.1005.2013.01265
[4] DICARLO J E, NORVILLE J E, MALI P, RIOS X, AACH J, CHURCH G M. Genome engineering in saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research, 2013, 41(7): 4336-4343. doi: 10.1093/nar/gkt135
[5] CONG L, RAN F A, COX D, LIN S L, BARRETTO R, HABIB N, HSU P D, WU X B, JIANG W Y, MARRAFFINI L A, ZHANG F. Multiplex genome engineering using CRISPR/Cas9 systems. Science, 2013, 339: 819-823. doi: 10.1126/science.1231143
[6] WANG H Y, YANG H, SHIVALILA C S, DAWLATY M M, CHENG A W, ZHANG F, JAENISCH R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas9-mediated genome engineering. Cell, 2013, 153(4): 910-918. doi: 10.1016/j.cell.2013.04.025
[7] JIANG W Z, ZHOU H B, BI H H, FROMM M, YANG B, WEEKS D P. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in arabidopsis, tobacco, sorghum and rice. Nucleic Acids Research, 2013, 41(20): e188. doi: 10.1093/nar/gkt780
[8] MAO Y, ZHANG H, XU N, ZHANG B, GOU F, ZHU J K. Application of the CRISPR-Cas system for efficient genome engineering in plants. Molecular Plant, 2013, 6(6): 2008-2011. doi: 10.1093/mp/sst121
[9] SHAN Q W, WANG Y P, LI J, ZHANG Y, CHEN K L, LIANG Z, ZHANG K, LIU J X, XI J J, QIU J L, GAO C X. Targeted genome modification of crop plants using a CRISPR-Cas system. Nature Biotechnology, 2013, 31(8): 686-688. doi: 10.1038/nbt.2650
[10] ZHANG H, ZHANG J S, WEI P L, ZHANG B T, GOU F, FENG Z Y, MAO Y F, YANG L, ZHANG H, XU N F, ZHU J K. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation. Plant Biotechnology Journal, 2014, 12(6): 797-807. doi: 10.1111/pbi.12200
[11] LI J F, NORVILLE J E, AACH J, MCCORMACK M, ZHANG D D, BUSH J, CHURCH G M, SHEEN J. Multiplex and homologous recombination-mediated genome editing in arabidopsis and nicotiana benthamiana using guide RNA and Cas9. Nature Biotechnology, 2013, 31(8): 688-691. doi: 10.1038/nbt.2654
[12] NEKRASOV V, STASKAWICZ B, WEIGEL D, JONES J D G, KAMOUN S. Targeted mutagenesis in the model plant nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nature Biotechnology, 2013, 31(8): 691-693. doi: 10.1038/nbt.2655
[13] LIANG Z, ZHANG K, CHEN K L, GAO C X. Targeted mutagenesis in zea mays using TALENs and the CRISPR/Cas9 system. Journal of Genetics and Genomics, 2014, 41(2): 63-68. doi: 10.1016/j.jgg.2013.12.001
[14] WANG Y P, CHENG X, SHAN Q W, ZHANG Y, LIU J X, GAO C X, QIU J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nature Biotechnology, 2014, 32(9): 947-951. doi: 10.1038/nbt.2969
[15] JACOBS T B, LAFAYETTE P R, SCHMITZ R J, PARROTT W A. Targeted genome modifications in soybean with CRISPR/Cas9. BMC Biotechnology, 2015, 15: 16. doi: 10.1186/s12896-015-0131-2
[16] LI Z S, LIU Z B, XING A Q, MOON B P, KOELLHOFFER J P, HUANG L X, WARD R T, CLIFTON E, FALCO S C, CIGAN A M. Cas9-guide RNA directed genome editing in soybean. Plant Physiology, 2015, 169(2): 960-970. doi: 10.1104/pp.15.00783
[17] MICHNO J M, WANG X B, LIU J Q, CURTIN S J, KONO T J, STUPAR R M. CRISPR/Cas9 mutagenesis of soybean and medicago truncatula using a new web-tool and a modified Cas9 enzyme. Gm Crops & Food-Biotechnology in Agriculture and the Food Chain, 2015, 6(4): 243-252.
[18] MENG Y Y, HOU Y L, WANG H, JI R H, LIU B, WEN J Q, NIU L F, LIN H. Targeted mutagenesis by CRISPR/Cas9 system in the model legume medicago truncatula. Plant Cell Reports, 2017, 36(2): 371-374. doi: 10.1007/s00299-016-2069-9
[19] CURTIN S J, TIFFIN P, GUHLIN J, TRUJILLO D I, BURGHARDT L T, ATKINS P, BALTES N J, DENNY R, VOYTAS D F, STUPAR R M, YOUNG N D. Validating genome-wide association candidates controlling quantitative variation in nodulation. Plant Physiology, 2017, 173(2): 921-931. doi: 10.1104/pp.16.01923
[20] PAN D, ZHANG L Q. Tandemly arrayed genes in vertebrate genomes. Comparative and Functional Genomics, 2008, 2008(3): 545269.
[21] FREELING M. Bias in plant gene content following different sorts of duplication: Tandem, whole-genome, segmental, or by transposition. Annual Review of Plant Biology, 2009, 60: 433-453. doi: 10.1146/annurev.arplant.043008.092122
[22] YOUNG N D, DEBELLE F, OLDROYD G E D, GEURTS R, CANNON S B, UDVARDI M K, BENEDITO V A, MAYER K F X, GOUZY J, SCHOOF H, VAN DE PEER Y, PROOST S, COOK D R, MEYERS B C, SPANNAGL M, CHEUNG F, DE MITA S, KRISHNAKUMAR V, GUNDLACH H, ZHOU S G, MUDGE J, BHARTI A K, MURRAY J D, NAOUMKINA M A, ROSEN B, SILVERSTEIN K A T, TANG H B, ROMBAUTS S, ZHAO P X, ZHOU P, BARBE V, BARDOU P, BECHNER M, BELLEC A, BERGER A, BERGES H, BIDWELL S, BISSELING T, CHOISNE N, COULOUX A, DENNY R, DESHPANDE S, DAI X B, DOYLE J J, DUDEZ A M, FARMER A D, FOUTEAU S, FRANKEN C, GIBELIN C, GISH J, GOLDSTEIN S, GONZALEZ A J, GREEN P J, HALLAB A, HARTOG M, HUA A, HUMPHRAY S J, JEONG D H, JING Y, JOCKER A, KENTON S M, KIM D J, KLEE K, LAI H S, LANG C T, LIN S P, MACMIL S L, MAGDELENAT G, MATTHEWS L, MCCORRISON J, MONAGHAN E L, MUN J H, NAJAR F Z, NICHOLSON C, NOIROT C, O'BLENESS M, PAULE C R, POULAIN J, PRION F, QIN B F, QU C M, RETZEL E F, RIDDLE C, SALLET E, SAMAIN S, SAMSON N, SANDERS I, SAURAT O, SCARPELLI C, SCHIEX T, SEGURENS B, SEVERIN A J, SHERRIER D J, SHI R H, SIMS S, SINGER S R, SINHAROY S, STERCK L, VIOLLET A, WANG B B, WANG K Q, WANG M Y, WANG X H, WARFSMANN J, WEISSENBACH J, WHITE D D, WHITE J D, WILEY G B, WINCKER P, XING Y B, YANG L M, YAO Z Y, YING F, ZHAI J X, ZHOU L P, ZUBER A, DENARIE J, DIXON R A, MAY G D, SCHWARTZ D C, ROGERS J, QUETIER F, TOWN C D, ROE B A. The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature, 2011, 480(7378): 520-524.
[23] CHOE S W, NOGUCHI T, FUJIOKA S, TAKATSUTO S, TISSIER C P, GREGORY B D, ROSS A S, TANAKA A, YOSHIDA S,TAX F E, FELDMANN K A. The arabidopsis dwf7/ste1 mutant is defective in the delta (7) sterol C-5 desaturation step leading to brassinosteroid biosynthesis. Plant Cell, 1999, 11(2): 207-221. doi: 10.1126/science.1138632
-
表 1 低氮培养基配方表
Table 1 Formula table of low nitrogen medium
组分 Component 母液浓度 Stock solution 终浓度 Final concentration 大量元素
Macro-element氯化钙 CaCl2 0.9 mol·L−1 0.9 mmol·L−1 七水硫酸镁 MgSO4 × 7H2O 0.5 mol·L−1 0.5 mmol·L−1 磷酸二氢钾 KH2PO4 0.7 mol·L−1 0.7 mmol·L−1 二水磷酸氢二钠 Na2HPO4 × 2H2O 0.4 mol·L−1 0.8 mmol·L−1 柠檬酸铁 Ferric citrate 0.02 mol·L−1 20 μmol·L−1 硝酸铵 NH4NO3 1 mol·L−1 1 mmol·L−1 微量元素
Micro-element氯化锰 MnCl2 1 mg·mL−1 50 μg·L−1 五水硫酸铜C uSO4 × 5H2O 1 mg·mL−1 100 μg·L−1 氯化锌 ZnCl2 1 mg·mL−1 50 μg·L−1 硼酸 H3BO3 1 mg·mL−1 100 μg·L−1 二水高锰酸钠 NaMnO4 × 2H2O 1 mg·mL−1 100 μg·L−1 琼脂 Agar 9 g·L−1 用氢氧化钾(KOH)溶液调节培养基pH = 7.4。
The medium pH was adjusted to 7.4 with potassium hydroxide (KOH) solution.表 2 转基因毛根检测所用引物
Table 2 Primer sequences for transgenic hair roots detection
引物 Primer 序列 Sequence G1-target1-F GAACCGTAAACCTGCACTAA G1-target1-R CACATCACTTTGCCTACGTC G1-target2-F CTGAGTACTTGACAGAAATC G1-target2-R CACTAATGTTGTGAACCGATG G2-target1-F GGAACGTTATCTTTGCTTC G2-target1-R CGAGCTTTGATCATGTAAAC G2-target2-F GGTAGAACTTGGCTGGACAA G2-target2-R GAGGTCCGTTAATGAACTGG G3-target1-F CTTGTGATATTCTCTTTTCT G3-target1-R ACGAAGAGTATCGAACATCC G3-target2-F GAGTACCTGACAGAAATTGG G3-target2-R TTAACTTCAACGTTCTACAG CAS9-F CACCAAGCACGTTGCTCAGA CAS9-R GAGAATCCTCCGGTCTGAAC -
[1] BELHAJ K, CHAPARRO-GARCIA A, KAMOUN S, PATRON N J, NEKRASOV V. Editing plant genomes with CRISPR/Cas9. Current Opinion in Biotechnology, 2015, 32(2): 76-84.
[2] BALTES N J, VOYTAS D F. Enabling plant synthetic biology through genome engineering. Trends in Biotechnology, 2015, 33(2): 120-131. doi: 10.1016/j.tibtech.2014.11.008
[3] JIANG W Y, BIKARD D, COX D, ZHANG F, MARRAFFINI L A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. doi: 10.3724/SP.J.1005.2013.01265
[4] DICARLO J E, NORVILLE J E, MALI P, RIOS X, AACH J, CHURCH G M. Genome engineering in saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research, 2013, 41(7): 4336-4343. doi: 10.1093/nar/gkt135
[5] CONG L, RAN F A, COX D, LIN S L, BARRETTO R, HABIB N, HSU P D, WU X B, JIANG W Y, MARRAFFINI L A, ZHANG F. Multiplex genome engineering using CRISPR/Cas9 systems. Science, 2013, 339: 819-823. doi: 10.1126/science.1231143
[6] WANG H Y, YANG H, SHIVALILA C S, DAWLATY M M, CHENG A W, ZHANG F, JAENISCH R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas9-mediated genome engineering. Cell, 2013, 153(4): 910-918. doi: 10.1016/j.cell.2013.04.025
[7] JIANG W Z, ZHOU H B, BI H H, FROMM M, YANG B, WEEKS D P. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in arabidopsis, tobacco, sorghum and rice. Nucleic Acids Research, 2013, 41(20): e188. doi: 10.1093/nar/gkt780
[8] MAO Y, ZHANG H, XU N, ZHANG B, GOU F, ZHU J K. Application of the CRISPR-Cas system for efficient genome engineering in plants. Molecular Plant, 2013, 6(6): 2008-2011. doi: 10.1093/mp/sst121
[9] SHAN Q W, WANG Y P, LI J, ZHANG Y, CHEN K L, LIANG Z, ZHANG K, LIU J X, XI J J, QIU J L, GAO C X. Targeted genome modification of crop plants using a CRISPR-Cas system. Nature Biotechnology, 2013, 31(8): 686-688. doi: 10.1038/nbt.2650
[10] ZHANG H, ZHANG J S, WEI P L, ZHANG B T, GOU F, FENG Z Y, MAO Y F, YANG L, ZHANG H, XU N F, ZHU J K. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation. Plant Biotechnology Journal, 2014, 12(6): 797-807. doi: 10.1111/pbi.12200
[11] LI J F, NORVILLE J E, AACH J, MCCORMACK M, ZHANG D D, BUSH J, CHURCH G M, SHEEN J. Multiplex and homologous recombination-mediated genome editing in arabidopsis and nicotiana benthamiana using guide RNA and Cas9. Nature Biotechnology, 2013, 31(8): 688-691. doi: 10.1038/nbt.2654
[12] NEKRASOV V, STASKAWICZ B, WEIGEL D, JONES J D G, KAMOUN S. Targeted mutagenesis in the model plant nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nature Biotechnology, 2013, 31(8): 691-693. doi: 10.1038/nbt.2655
[13] LIANG Z, ZHANG K, CHEN K L, GAO C X. Targeted mutagenesis in zea mays using TALENs and the CRISPR/Cas9 system. Journal of Genetics and Genomics, 2014, 41(2): 63-68. doi: 10.1016/j.jgg.2013.12.001
[14] WANG Y P, CHENG X, SHAN Q W, ZHANG Y, LIU J X, GAO C X, QIU J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nature Biotechnology, 2014, 32(9): 947-951. doi: 10.1038/nbt.2969
[15] JACOBS T B, LAFAYETTE P R, SCHMITZ R J, PARROTT W A. Targeted genome modifications in soybean with CRISPR/Cas9. BMC Biotechnology, 2015, 15: 16. doi: 10.1186/s12896-015-0131-2
[16] LI Z S, LIU Z B, XING A Q, MOON B P, KOELLHOFFER J P, HUANG L X, WARD R T, CLIFTON E, FALCO S C, CIGAN A M. Cas9-guide RNA directed genome editing in soybean. Plant Physiology, 2015, 169(2): 960-970. doi: 10.1104/pp.15.00783
[17] MICHNO J M, WANG X B, LIU J Q, CURTIN S J, KONO T J, STUPAR R M. CRISPR/Cas9 mutagenesis of soybean and medicago truncatula using a new web-tool and a modified Cas9 enzyme. Gm Crops & Food-Biotechnology in Agriculture and the Food Chain, 2015, 6(4): 243-252.
[18] MENG Y Y, HOU Y L, WANG H, JI R H, LIU B, WEN J Q, NIU L F, LIN H. Targeted mutagenesis by CRISPR/Cas9 system in the model legume medicago truncatula. Plant Cell Reports, 2017, 36(2): 371-374. doi: 10.1007/s00299-016-2069-9
[19] CURTIN S J, TIFFIN P, GUHLIN J, TRUJILLO D I, BURGHARDT L T, ATKINS P, BALTES N J, DENNY R, VOYTAS D F, STUPAR R M, YOUNG N D. Validating genome-wide association candidates controlling quantitative variation in nodulation. Plant Physiology, 2017, 173(2): 921-931. doi: 10.1104/pp.16.01923
[20] PAN D, ZHANG L Q. Tandemly arrayed genes in vertebrate genomes. Comparative and Functional Genomics, 2008, 2008(3): 545269.
[21] FREELING M. Bias in plant gene content following different sorts of duplication: Tandem, whole-genome, segmental, or by transposition. Annual Review of Plant Biology, 2009, 60: 433-453. doi: 10.1146/annurev.arplant.043008.092122
[22] YOUNG N D, DEBELLE F, OLDROYD G E D, GEURTS R, CANNON S B, UDVARDI M K, BENEDITO V A, MAYER K F X, GOUZY J, SCHOOF H, VAN DE PEER Y, PROOST S, COOK D R, MEYERS B C, SPANNAGL M, CHEUNG F, DE MITA S, KRISHNAKUMAR V, GUNDLACH H, ZHOU S G, MUDGE J, BHARTI A K, MURRAY J D, NAOUMKINA M A, ROSEN B, SILVERSTEIN K A T, TANG H B, ROMBAUTS S, ZHAO P X, ZHOU P, BARBE V, BARDOU P, BECHNER M, BELLEC A, BERGER A, BERGES H, BIDWELL S, BISSELING T, CHOISNE N, COULOUX A, DENNY R, DESHPANDE S, DAI X B, DOYLE J J, DUDEZ A M, FARMER A D, FOUTEAU S, FRANKEN C, GIBELIN C, GISH J, GOLDSTEIN S, GONZALEZ A J, GREEN P J, HALLAB A, HARTOG M, HUA A, HUMPHRAY S J, JEONG D H, JING Y, JOCKER A, KENTON S M, KIM D J, KLEE K, LAI H S, LANG C T, LIN S P, MACMIL S L, MAGDELENAT G, MATTHEWS L, MCCORRISON J, MONAGHAN E L, MUN J H, NAJAR F Z, NICHOLSON C, NOIROT C, O'BLENESS M, PAULE C R, POULAIN J, PRION F, QIN B F, QU C M, RETZEL E F, RIDDLE C, SALLET E, SAMAIN S, SAMSON N, SANDERS I, SAURAT O, SCARPELLI C, SCHIEX T, SEGURENS B, SEVERIN A J, SHERRIER D J, SHI R H, SIMS S, SINGER S R, SINHAROY S, STERCK L, VIOLLET A, WANG B B, WANG K Q, WANG M Y, WANG X H, WARFSMANN J, WEISSENBACH J, WHITE D D, WHITE J D, WILEY G B, WINCKER P, XING Y B, YANG L M, YAO Z Y, YING F, ZHAI J X, ZHOU L P, ZUBER A, DENARIE J, DIXON R A, MAY G D, SCHWARTZ D C, ROGERS J, QUETIER F, TOWN C D, ROE B A. The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature, 2011, 480(7378): 520-524.
[23] CHOE S W, NOGUCHI T, FUJIOKA S, TAKATSUTO S, TISSIER C P, GREGORY B D, ROSS A S, TANAKA A, YOSHIDA S,TAX F E, FELDMANN K A. The arabidopsis dwf7/ste1 mutant is defective in the delta (7) sterol C-5 desaturation step leading to brassinosteroid biosynthesis. Plant Cell, 1999, 11(2): 207-221. doi: 10.1126/science.1138632
-
期刊类型引用(27)
1. 刘祥年,殷熙彤,唐于然,王鹏飞,滚双宝. 不同比例全株青贮玉米对合作猪屠宰性能、器官指数、肉品质和经济效益的影响. 饲料工业. 2025(01): 64-69 .  百度学术
百度学术
2. 贾海阔,吴春会,祖晓伟,刘振宇,陈欢,刘震灵,王明亚,李秋凤. 不同添加剂和贮藏时间对苜蓿青贮品质和霉菌数量的影响. 饲料研究. 2025(01): 163-169 .  百度学术
百度学术
3. 谢欣杰,郑伟华,李东健,李耀,李凤鸣. 不同水分和发酵时间对番茄渣和麦秸混贮效果的影响. 饲料工业. 2024(01): 17-20 .  百度学术
百度学术
4. 高燕春,袁凯,刘爱菊. 不同微生物发酵剂对青贮玉米营养成分、感官评定及发酵品质的影响. 中国饲料. 2024(02): 76-79 .  百度学术
百度学术
5. 陆龙超,莫本田,周文章,秦杨,邓似辰,王清峰,徐龙鑫,武俊达. 菌酶添加对喀斯特地区全株青贮玉米发酵品质和有氧稳定性的影响. 饲料工业. 2024(03): 95-101 .  百度学术
百度学术
6. 代友超,马越,徐美玲,房华,王瑛,贾莉,史金平,雷赵民. 玉米品种和发酵剂对全株玉米青贮营养品质的影响. 动物营养学报. 2024(01): 658-671 .  百度学术
百度学术
7. 何晓琳,王胜男,肖艺梅,尹福泉,卢艳丽. 收获时间与留茬高度对桂青贮7号青贮玉米体外消化特性的影响. 动物营养学报. 2024(02): 1315-1330 .  百度学术
百度学术
8. 李旭业,董扬,尤海洋,郭文凯,王佳. 不同类型添加剂对玉米秸秆青贮品质的影响. 饲料研究. 2024(05): 99-103 .  百度学术
百度学术
9. 周小栋,马成艳,张嘉琦,黄帅. 柠条叶提取物对苜蓿青贮品质及抗氧化活性的影响. 草业科学. 2024(04): 995-1006 .  本站查看
本站查看
10. 潘晓辉. 反刍动物对饲草不同加工产品高效利用的研究进展. 饲料博览. 2024(01): 20-24 .  百度学术
百度学术
11. 吴鹏鑫,王胜男,杨伟光,王志敬,尹福泉. 收获期对冬种甜玉米秸秆青贮品质的影响. 饲料研究. 2024(08): 114-120 .  百度学术
百度学术
12. 吉方财,李雨嘉,张洪瑞,王一凡,王磊,卓兴良,孙志强,寇启芳,玉柱. 不同收获期与添加剂处理对甜高粱青贮品质及体外消化率的影响. 动物营养学报. 2024(05): 3373-3385 .  百度学术
百度学术
13. 左愈臻,刘瑞刚,魏旭辉,汪旭. 不同青贮发酵促进剂和青贮时间对全株青贮玉米品质的影响. 畜牧兽医杂志. 2024(05): 22-28 .  百度学术
百度学术
14. 唐云会,鲁茸格列,周宇斌,尹亚敏,罗富成. 水分含量对杂交臂形草青贮品质的影响. 饲料研究. 2024(09): 118-121 .  百度学术
百度学术
15. 左愈臻,刘瑞刚,魏旭辉,汪旭. 不同青贮发酵促进剂对全株青贮玉米发酵品质的影响. 甘肃畜牧兽医. 2024(03): 73-76 .  百度学术
百度学术
16. 魏椿萱,达朝鹏,李聪. 饲喂全株玉米青贮饲料对肉牛养殖的影响. 现代畜牧科技. 2024(07): 64-66 .  百度学术
百度学术
17. 左愈臻,魏旭辉,刘瑞刚,汪旭. 不同青贮发酵促进剂对全株青贮玉米营养成分的影响. 甘肃畜牧兽医. 2024(04): 70-73 .  百度学术
百度学术
18. 王艳霞. 不同添加剂对紫花苜蓿青贮饲料感官品质、营养成分及发酵品质的影响. 中国饲料. 2024(18): 89-92 .  百度学术
百度学术
19. 左愈臻,刘瑞刚,魏旭辉,汪旭. 不同青贮发酵促进剂和青贮时间对全株青贮玉米品质的影响. 畜牧兽医杂志. 2024(03): 22-28 .  百度学术
百度学术
20. 兰英,赵秀梅,刘悦,李青超,王立达,刘洋. 杀菌剂在青贮玉米生产中的应用及研究进展. 园艺与种苗. 2024(11): 93-95 .  百度学术
百度学术
21. 张洪瑞,吉方财,王一凡,苑明伟,陈雪,刘奕婷,王磊,包锦泽,孙志强,玉柱. 不同栽培措施对全株玉米青贮效果及体外消化率的影响. 草业科学. 2024(12): 3016-3024 .  本站查看
本站查看
22. 鲍俊杰,郭广振,凡超杰,杨伟光,卢艳丽,尹福泉. 基于主成分分析和隶属函数分析研究收获期与青贮对甜玉米秸秆饲用品质的影响. 动物营养学报. 2023(04): 2677-2689 .  百度学术
百度学术
23. 王诚,赵洪伟,范秋苹,董桂红,何荣彦,翟桂玉,刘德娟. 收获时间和留茬高度对全株玉米青贮品质及营养成分的影响. 现代畜牧兽医. 2023(06): 24-27 .  百度学术
百度学术
24. 李春芳,崔秋佳,王栓柱,刘鑫雅,侯秀贤,杨晨东,蒋桂娥,赵利梅,马亚宾,李建明. 2019-2021年河北省全株玉米青贮品质对比研究. 中国奶牛. 2023(08): 56-61 .  百度学术
百度学术
25. 高永权,焦婷,杨永慧,路平乐,王虎宁,李淑艳,齐帅. 庆阳市3个肉牛养殖场青贮玉米品质评价. 草原与草坪. 2023(06): 137-144 .  百度学术
百度学术
26. 张晓,苏亚军,王瑛,范凯利,孙建祥,张瑞,贾莉,雷赵民. 品种和密度对青贮玉米生物量与农艺性状的影响. 饲料研究. 2022(15): 88-93 .  百度学术
百度学术
27. 孙浩,卢家顶,史莹华,王成章. 全株小麦青贮在动物生产中的应用及前景. 草业科学. 2022(11): 2453-2465 .  本站查看
本站查看
其他类型引用(5)



 下载:
下载: