紫花苜蓿ADK基因家族成员鉴定与分析
English
-
腺苷酸代谢不仅是生命活动中初级代谢的重要组成部分,也是影响细胞代谢的主要因素之一。核酸代谢库前体分子单磷酸腺苷(adenosine monophosphate, AMP)是构成细胞核糖核酸的主要单核苷酸之一,AMP的形成通常伴随着生物体内能量的释放。AMP、能量分子二磷酸腺苷(adenosine diphosphate, ADP)和糖类代谢库前体三磷酸腺苷(adenosine triphosphate, ATP) 3种形式的比例决定了能量电荷比和碳水化合物代谢,进而直接影响植物的生长发育和对外界不良环境的适应性[1-2]。腺苷酸激酶(adenylate kinase, ADK, EC 2.7.4.3)是维持能量代谢和各种腺苷酸库大小平衡的单体磷酸转移酶,催化ATP + AMP ↔ 2ADP的可逆转磷酸化反应[3-4]。在植物线粒体中,ADK的活性将会显著影响细胞质和质体中游离态与结合态镁离子在腺苷酸库中的代谢平衡。
通常,ADK蛋白由一个AMP结构域,一个ATP结构域和一个相对保守的核心结构域(CORE)组成[5]。研究表明,ADK在动植物中高度保守,其活性已在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、玉米(Zea mays)、豌豆(Pisum sativum)、马铃薯(Solanum tuberosum)等植物中得到证实,而ADK的亚细胞定位在不同植物中差异很大,如在细胞质、线粒体、细胞核、质体中等均有报道[6-10]。在拟南芥中,鉴定到一个重要的茎生长调控因子AAK6,T-DNA插入纯合突变体(Salk_015281)与野生型植株相比,突变体茎的生长减缓[11];拟南芥ADK (At2g37250)基因的T-DNA插入纯合突变体,表现出氨基酸含量提高和根生长增强的特性[12];另一项相关研究表明,拟南芥ADK (At5g47840)基因的T-DNA插入纯合突变体,植株叶绿体完整性丧失,从而导致早期胚胎到幼苗发育过程中出现白化苗,表明ADK基因在拟南芥的生长发育过程中发挥重要调控作用[13]。在马铃薯中的研究表明,沉默StADK基因的表达后,转基因植株的腺苷酸含量和淀粉含量得到显着提高[14]。此外,ADK3可以与叶绿体3−磷酸甘油醛脱氢酶相互作用,在绿藻(Chlorophyta)的叶绿体中形成稳定的复合物[15]。除了在调控植物生长和发育方面发挥重要功能,ADK还广泛参与植物对非生物胁迫的应激反应。以豌豆种子为模型,研究了种子脱水吸水过程中腺苷酸的平衡情况,结果表明ADK的活性在保持种子干燥和成熟过程中腺苷酸的平衡中发挥关键作用[16]。大豆(Glycine max) GmADK基因受到盐胁迫后,在耐盐品种中的表达高于盐敏感性品种,推测大豆GmADK基因参与盐胁迫的应答[17]。然而在番茄(Solanum lycopersicum)中,基因的微阵列分析显示,ADK同源基因(SGN-U214214)在盐处理的番茄组织中表达受到抑制,在耐旱番茄品种中,ADK基因(SGN-U232826)的表达受到干旱胁迫的诱导[18]。目前,已在拟南芥、番茄和水稻中分别鉴定到7、10和11个ADK基因,不同成员间的亚细胞定位,表达模式存在差异,具体的生理功能还需进一步研究[19-20]。虽然ADK基因在生物体能量代谢中发挥重要功能,但是在豆科牧草中的研究尚未见报道。
紫花苜蓿(Medicago sativa)是奶牛等草食动物的重要优质饲草,素有“牧草之王”的美誉[21]。近年来,国家实施振兴奶业苜蓿发展行动,有力促进了苜蓿产业的发展。尽管已在其他植物中鉴定、分析了ADK基因家族,但是紫花苜蓿ADK基因的功能仍有待进一步阐明。在本研究中,利用生物信息学方法从紫花苜蓿基因组中鉴定了ADK基因,并分析了序列特征、基因结构、染色体定位、进化关系、顺式作用元件,以及ADK基因在不同组织和非生物胁迫下[干旱、低温、盐和脱落酸(abscisic acid,ABA)]的表达模式。研究结果将为进一步研究MsADK家族的功能和调控机制提供有用信息。
1. 材料与方法
1.1 紫花苜蓿ADK基因家族鉴定
紫花苜蓿基因组序列、蛋白质序列、CDS序列,以及gff文件下载自figshare数据库(https://figshare.com/projects/whole_genome_sequencing_and_assembly_of_Medicago_sativa/66380)。ADK蛋白的保守结构域序列(PF00406)下载自pfam数据库(http://pfam.xfam.org/),在bio-linux系统下通过“Hmmsearch”命令鉴定紫花苜蓿ADK家族成员,并在CDD数据库(https://www.ncbi.nlm.nih.gov/cdd/)进一步验证是否含有ADK蛋白的保守结构域。最后,利用CD-HIT (http://weizhong-lab.ucsd.edu/cd-hit/)软件去除冗余序列,序列特征参数为98%。
1.2 紫花苜蓿ADK蛋白质理化性质,亚细胞定位,二级和三级结构预测分析
利用在线工具ProtParam (https://web.expasy.org/protparam/)预测MsADK蛋白质的氨基酸数、分子量、等电点、疏水性指数、稳定指数等理化性质。在WoLF PSORT II网站(https://www.genscript.com/wolf-psort.html)预测MsADK蛋白成员的亚细胞定位。通过在线工具SOMPA (https://prabi.ibcp.fr/htm/site/web/home)预测MsADK蛋白质二级结构,SWISS-MODE (https://swissmodel.expasy.org/)预测MsADK蛋白质三级结构。
1.3 紫花苜蓿ADK基因系统进化树分析
利用MEGA 7软件构建34个ADK蛋白序列的系统进化树,包括紫花苜蓿(10个MsADK)、拟南芥(7个AtADK)、蒺藜苜蓿(Medicago truncatula,7个MtADK)和水稻(10个OsADK)。采用最大似然法(maximum likelihood estimate, MLE),步长值(bootstrap values)为1 000次,其他参数设置默认。
1.4 紫花苜蓿ADK基因结构、保守基序和顺式作用原件分析
GSDS 2. 0 (http://gsds.gao-lab.org/index.php)工具用于分析MsADK成员的基因结构。MEME (https://meme-suite.org/meme/tools/meme)用于预测蛋白的保守基序,保守基序数为10,其他参数默认。PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)工具用于启动子序列(2.0 kb)顺式作用原件分析,并通过TBtools工具进行可视化分析[22]。
1.5 紫花苜蓿ADK基因共线性和染色体定位分析
在Phytozome 13 (https://phytozome-next.jgi.doe.gov/)网站分别下载拟南芥、蒺藜苜蓿以及大豆的全基因组序列和gff文件,利用TBtools工具对MsADK基因成员进行共线性、基因重复事件和染色体定位分析,并实现结果的可视化。
1.6 紫花苜蓿ADK基因的表达模式分析
利用BLAST比对工具在Alfalfa Breeder’s Toolbox (https://www.alfalfatoolbox.org/)数据库鉴定MsADK最匹配的转录本序列,得到MsADK基因分别在紫花苜蓿和黄花苜蓿中不同组织的表达量[23]。通过本地BLAST程序检索紫花苜蓿‘中苜1号’的转录本序列,获取MsADK基因分别在干旱、低温、ABA以及盐胁迫下的表达模式[24-26],并利用TBtools工具绘制热图。
2. 结果与分析
2.1 紫花苜蓿ADK基因家族成员信息
紫花苜蓿基因组中共鉴定到10个MsADK基因,并按照染色体上排序命名为MsADK1-MsADK10 (表1)。MsADK蛋白的平均长度为273.4 aa,其中MsADK2最长(504 aa),其余序列的长度均在250 aa左右。MsADK蛋白的平均分子量30.5 kDa,其中MsADK2的分子量最大为56.8 kDa,其余蛋白均小于31 kDa;蛋白质理论等电点介于5.46 (MsADK10)~9.33 (MsADK8)。MsADK蛋白的疏水性指数(GRAVY)的平均值为−0.31,均小于0,表明MsADK为亲水性蛋白。亚细胞定位预测表明,MsADK2、MsADK5、MsADK9和MsADK10定位于细胞质;MsADK1和MsADK3定位于叶绿体;MsADK7和MsADK8定位于线粒体;MsADK4定位于过氧化体;MsADK6定位于细胞核。除MsADK9 (Ⅱ = 35.91)蛋白的稳定指数小于40,表现为蛋白稳定外,其余MsADK蛋白均表现为不稳定。与拟南芥基因组序列进行BLAST比对,结果表明,MsADK包含了拟南芥中全部的ADK同源基因。
表 1 紫花苜蓿ADK基因家族信息Table 1. Information of ADK gene family in Medicago sativa基因
名称
Gene
Name基因号
Gene
ID染色体
Chr.
No蛋白
长度
Amino acids
length/aacDNA
长度
cDNA
length/bp蛋白质
分子量
Protein molecular
weight/kDa等电
点
pI疏水性
指数
Protein
GRAVY亚细胞
定位
Subcellular
localization蛋白稳定
指数
Instability
index拟南芥
同源基因
Homologous gene
of ArabidopsisMsADK1 MS.gene029884.t1 chr1.4 280 843 30.75 6.67 −0.164 叶绿体
Chloroplast44.68 AT2G37250.1 MsADK2 MS.gene81523.t1 chr2.2 504 1515 56.79 6.41 −0.382 细胞质
Cytoplasmic42.45 AT5G35170.1 MsADK3 MS.gene045674.t1 chr4.3 244 735 27.71 8.64 −0.312 叶绿体
Chloroplast49.23 AT4G25280.2 MsADK4 MS.gene77729.t1 chr4.4 242 729 27.52 7.29 −0.324 过氧化体
Peroxisomal52.32 AT4G25280.2 MsADK5 MS.gene97107.t1 chr4.4 234 705 26.11 6.53 −0.311 细胞质
Cytoplasmic35.63 AT5G47840.1 MsADK6 MS.gene060445.t1 chr7.1 265 798 29.90 6.80 −0.448 细胞核
Nuclear50.29 AT3G01820.1 MsADK7 MS.gene007478.t1 chr7.1 268 807 219.28 7.39 −0.206 线粒体
Mitochondrial53.86 AT2G37250.1 MsADK8 MS.gene09796.t1 chr7.3 246 738 27.04 9.33 −0.150 线粒体
Mitochondrial48.54 AT2G37250.1 MsADK9 MS.gene011389.t1 chr8.3 243 732 26.67 7.97 −0.326 细胞质
Cytoplasmic35.91 AT5G63400.1 MsADK10 MS.gene39664.t1 chr8.3 208 627 23.15 5.46 −0.466 细胞质
Cytoplasmic43.44 AT5G26667.4 pI:等电点; GRAVY:疏水性指数。
pI: isoelectric; GRAVY: grand average of hydropathicity.2.2 紫花苜蓿ADK家族成员系统进化分析
MsADK与拟南芥(双子叶)、水稻(单子叶)、蒺藜苜蓿(豆科) ADK的进化关系表明,10个MsADK被划分为5个(Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ)亚家族(图1)。每个亚家族均包含上述4个物种的ADK成员,表明ADK在不同物种间高度保守,在单双子叶物种分离前就已存在。MsADK蛋白与蒺藜苜蓿ADK蛋白高度同源,拟南芥次之,水稻最远。紫花苜蓿MsADK数目大于蒺藜苜蓿,其中MsADK6和MsADK10被划分为单独的分枝,说明ADK在紫花苜蓿中更为复杂。在Ⅰ亚家族中包含MsADK1、MsADK7和MsADK8;在Ⅱ亚家族中包含MsADK2和MsADK5两个成员;在Ⅲ亚家族中仅有MsADK9;在Ⅳ亚家族中包含MsADK3、MsADK4和MsADK10;在Ⅴ亚家族中包含MsADK6一个成员。
![]() 图 1 紫花苜蓿、蒺藜苜蓿、拟南芥和水稻ADK蛋白的系统进化分析图中正方形、三角形、圆形和菱形分别代表紫花苜蓿、水稻、蒺藜苜蓿和拟南芥。Figure 1. Phylogenetic tree of ADK genes from Medicago sativa, M. truncatula, Arabidopsis thaliana, and Oryza sativaSquares, triangles, circles and diamonds represent Medicago sativa, Oryza sativa, M. truncatula, and, Arabidopsis thaliana, respectively.
图 1 紫花苜蓿、蒺藜苜蓿、拟南芥和水稻ADK蛋白的系统进化分析图中正方形、三角形、圆形和菱形分别代表紫花苜蓿、水稻、蒺藜苜蓿和拟南芥。Figure 1. Phylogenetic tree of ADK genes from Medicago sativa, M. truncatula, Arabidopsis thaliana, and Oryza sativaSquares, triangles, circles and diamonds represent Medicago sativa, Oryza sativa, M. truncatula, and, Arabidopsis thaliana, respectively.2.3 紫花苜蓿ADK保守基序和基因结构分析
依据MsADK基因的进化距离进行基因结构和保守基序分析(图2),结果表明,MsADK蛋白的10个基序(Motif 1~Motif 10)在不同亚家族中分布具有差异,每个基因都含有4~7个保守基序不等。MsADK蛋白均含有Motif 1和Motif 3两个基序,表明这两个基序是MsADK家族的核心组成。Motif 4和Motif 7仅在Ⅰ和Ⅴ亚家族中存在;Motif 9仅在Ⅱ和Ⅲ亚家族中存在;Motif 8仅在Ⅳ和Ⅴ亚家族中存在;不同亚家族特定的基序可能决定了MsADK基因的功能多样性。MsADK基因的外显子-内含子结构图表明(图2B),各成员间外显子数目4~18个,以3~6个居多(60%),MsADK2基因序列最长,含外显子数目最多为18个。
2.4 紫花苜蓿MsADK基因染色体定位和共线性分析
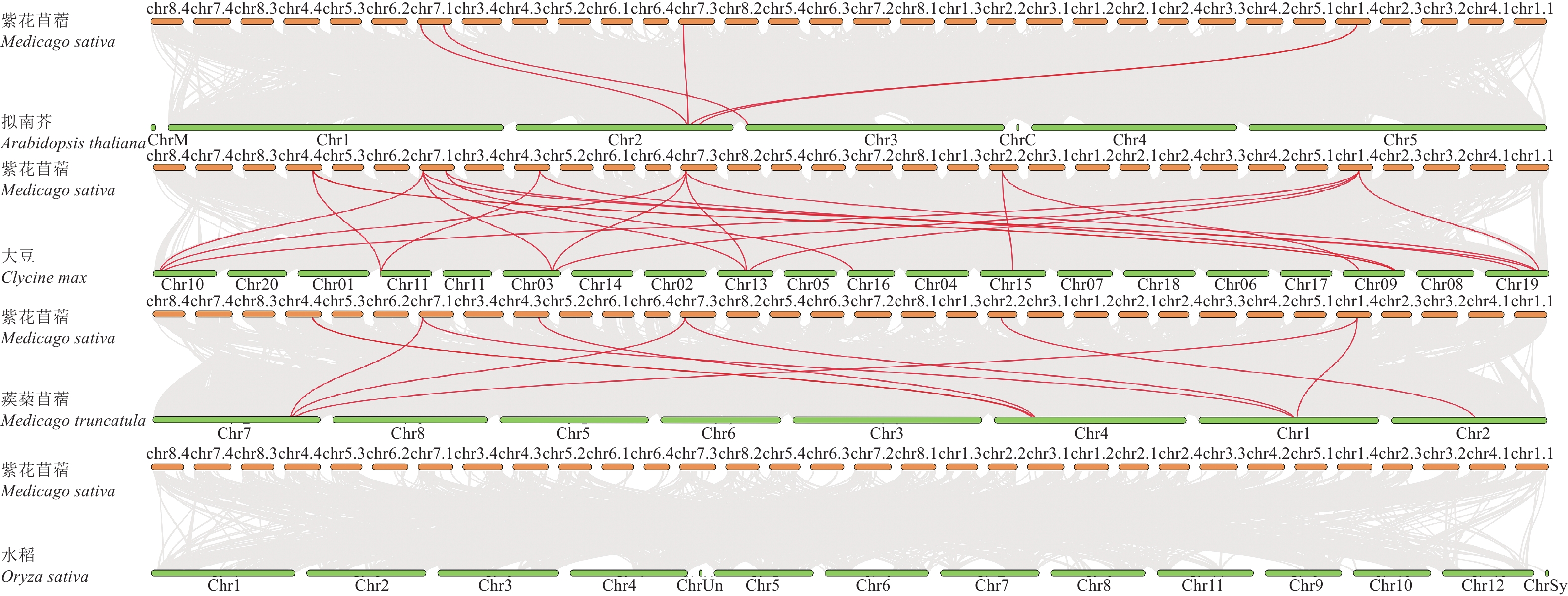
对10个MsADK基因进行染色体定位分析,家族成员随机的分布在32条同源染色体中的7条上(图3)。大多数MsADK基因位于染色体的近端或远端,chr4.4、chr7.1和chr8.3染色体上均有两个MsADK基因;chr1.4、chr2.2、chr4.3和chr7.3染色体上均只有1个MsADK基因。对MsADK基因进行共线性分析,基因对MsADK1/MsADK7、MsADK3/MsADK4和MsADK7/MsADK8之间存在共线性关系,并被划分到同一亚类,推断上述3对基因是经过全基因组复制而形成的。
为了进一步推断ADK基因成员的系统发育机制,分别构建了MsADK基因与拟南芥、水稻、蒺藜苜蓿和大豆4个代表性物种的比较共线性图谱,其中MsADK基因和水稻基因间不存在共线性关系(图4)。紫花苜蓿和大豆,蒺藜苜蓿都属于豆科植物,同源性更近。MsADK基因与21个大豆间存在共线性关系(MsADK7/Glyma.03G176800/Glyma.10G048200/Glyma.13G136000/Glyma.19G177500、MsADK1/Glyma.10G048200/Glyma.03G176800/Glyma.13G136000/Glyma.19G177500、MsADK3/Glyma.12G004200/Glyma.09G232300、MsADK6/Glyma.19G088000/Glyma.16G059400、MsADK8/Glyma.10G048200/Glyma.03G176800/Glyma.13G136000/Glyma.19G177500、MsADK4/Glyma.09G232300/Glyma.12G004200、MsADK2/Glyma.15G203100/Glyma.09G096800、MsADK5/Glyma.09G234300);10个MsADK基因与蒺藜苜蓿间存在共线性关系(MsADK7/Medtr7g100530/Medtr1g067050、MsADK1/Medtr7g100530/Medtr1g067050、MsADK3/Medtr4g035850、MsADK8/Medtr7g100530/Medtr1g067050、MsADK4/Medtr4g035850、MsADK2/Medtr2g461290、和MsADK5/Medtr4g035170);5个MsADK基因与拟南芥基因存在共线性关系(MsADK1/AT2G37250/AT2G37250、MsADK6/AT3G01820、MsADK8/AT2G37250、MsADK7/AT2G37250)。
2.5 紫花苜蓿ADK基因启动子顺式作用元件分析
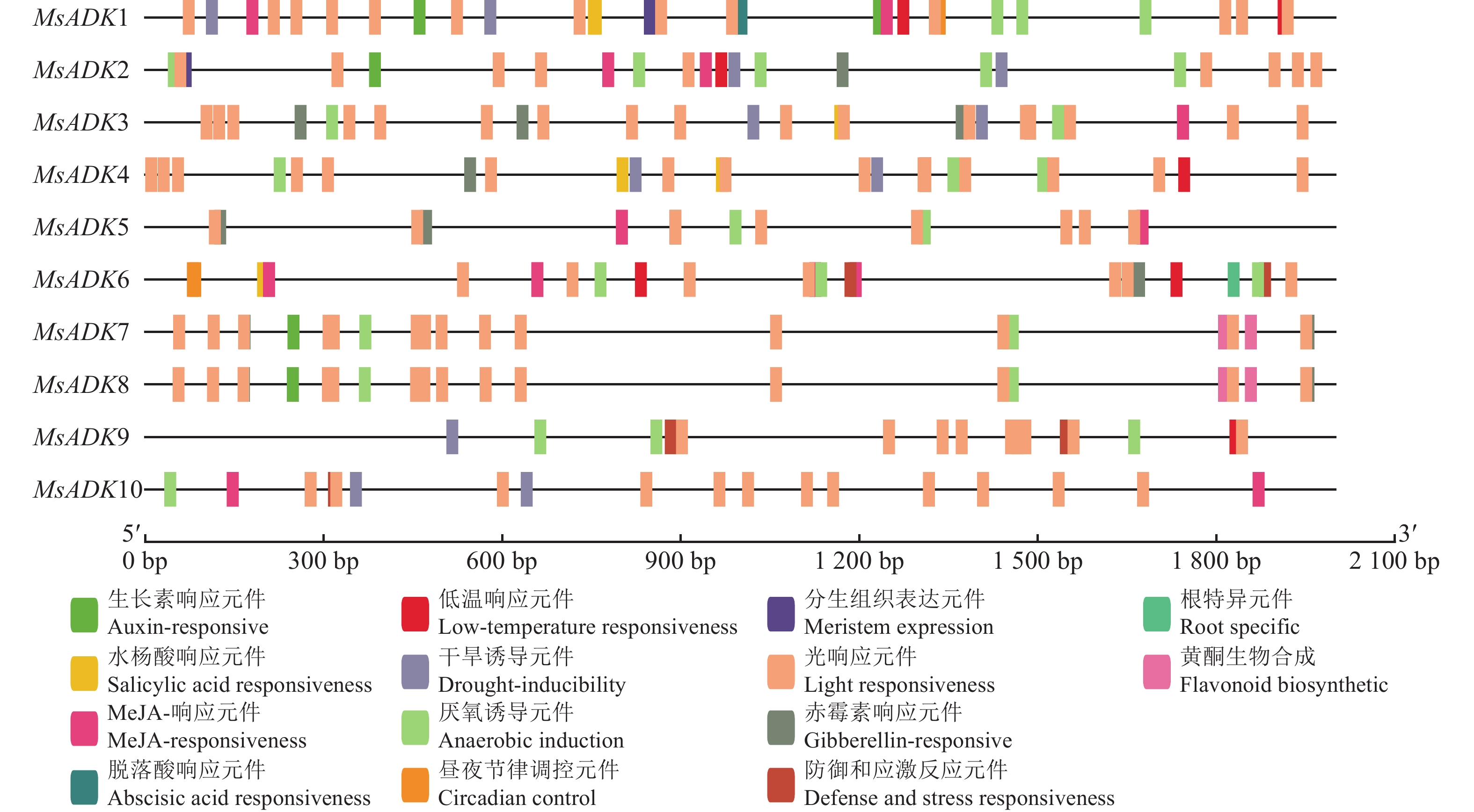
MsADK基因顺式作用元件预测分析结果显示(图5),上游2.0 kb的启动子序列中含有大量与激素和生长发育(如植物激素、非生物胁迫、光照、生理生长等)有关的作用元件,但是成员间含有的顺式元件的类型和数量均有所差异。其中MsADK1和MsADK6所含元件种类最多为10种。所有MsADK家族成员的启动子序列中都含有与植物激素相关的元件,如MsADK1、MsADK2、MsADK6、MsADK7、MsADK8含有生长素(TGA-element、TGA-box)响应元件;MsADK2、MsADK3、MsADK4、MsADK5、MsADK6、MsADK7、MsADK8含有赤霉素(P-box、TATC-box、GARE-motif)响应元件;MsADK1、MsADK3、MsADK4、MsADK5、MsADK7、MsADK8、MsADK9含有脱落酸(abscisic acid responsiveness,ABRE)响应元件;MsADK1、MsADK3、MsADK4、MsADK6含有水杨酸(salicylic acid responsiveness,SARE和TCA-element)响应元件;MsADK1、MsADK2、MsADK3、MsADK5、MsADK6、MsADK10含有茉莉酸(CGTCA-motif和TGACG-motif)响应元件。响应胁迫相关作用元件在MsADK家族中同样分布比较广泛,所有基因均含有厌氧诱导元件(anaerobic induction,ARE)和光响应元件(AAAC-motif、ACE、AE-box、AT1-motif、ATC-motif、ATCT-motif、Box 4、chs-CMA1a、chs-CMA2a、GA-motif、Gap-box、GATA-motif、G-box、GT1-motif、GTGGC-motif、I-box、LAMP-element、MRE、TCCC-motif、TCT-motif),MsADK6、MsADK9、MsADK10含有防御与应激响应元件(TC-rich repeats),MsADK1、MsADK2、MsADK3、MsADK4、MsADK9、MsADK10含有干旱诱导元件(MYB binding site involved in drought-inducibility,MBS);MsADK1、MsADK2、MsADK4、MsADK6含有低温响应元件(low temperature responsiveness,LTR);此外,启动子区域还有与生长发育和合成代谢相关的作用元件,如昼夜节律调控(Circadian)、黄酮生物合成(MYB binding site involved in flavonoid biosynthesis,MBSI)、分生组织表达(GCN4_motif)和根特异性(Motif I)等,表明MsADK同样在紫花苜蓿的生长发育过程中发挥重要功能。
2.6 MsADK蛋白二级、三级结构预测
蛋白质结构的变化将会直接影响其功能的改变,SOMPA预测结果显示,MsADK蛋白的蛋白二级结构以α−螺旋和无规则卷曲为主,β−转角和延伸链所占比例较低。蛋白质的α−螺旋占37.40%~53.72%,平均值为46.43%;无规则卷曲占27.35%~45.93%,平均值为33.89%;延伸链占10.71%~12.98%,平均值为11.84%;β−转角绝大多数低于10%,介于5.69%~10.29%,平均值为8.24% (表2)。推测MsADK蛋白序列的二级结构中,α−螺旋和无规则卷曲起主要作用。
表 2 MsADK蛋白二级结构Table 2. Secondary structure of MsADK proteins基因名称
Gene nameα-螺旋 α-helix β-转角 β-turn 无规则卷曲 Random coil 延伸链 Extended chain 数量
Number占比
Ratio/%数量
Number占比
Ratio/%数量
Number占比
Ratio/%数量
Number占比
Ratio/%MsADK1 125 44.64 27 9.64 98 35.00 30 10.71 MsADK2 213 46.26 41 8.13 180 35.71 70 13.89 MsADK3 113 46.31 16 6.56 84 34.43 31 12.70 MsADK4 130 53.72 18 7.44 67 27.69 27 11.16 MsADK5 121 51.71 21 8.97 64 27.35 28 11.97 MsADK6 134 50.57 21 7.92 79 29.81 31 11.70 MsADK7 111 41.42 23 8.58 105 39.18 29 10.82 MsADK8 92 37.40 14 5.69 113 45.93 27 10.98 MsADK9 111 45.68 25 10.29 79 32.51 28 11.52 MsADK10 97 46.63 19 9.13 65 31.25 27 12.98 平均值 Mean 124.7 46.43 22.5 8.24 93.4 33.89 32.8 11.84 研究表明,不同种属ADK蛋白的三维结构具有相同的,由α/β折叠构成的“三明治”状结构。利用SWISS-MODEL在线工具预测MsADK蛋白的三级结构,MsADK蛋白都含有α−螺旋、β−折叠、无规则卷曲等空间构象,MsADK6和MsADK7构象最为接近,其余同成员间结构具有差异性(图6)。另外,各MsADK成员含有的螺旋−转角−螺旋(HTH)数目存在差异。
2.7 紫花苜蓿ADK基因在不同组织和非生物胁迫下的表达分析
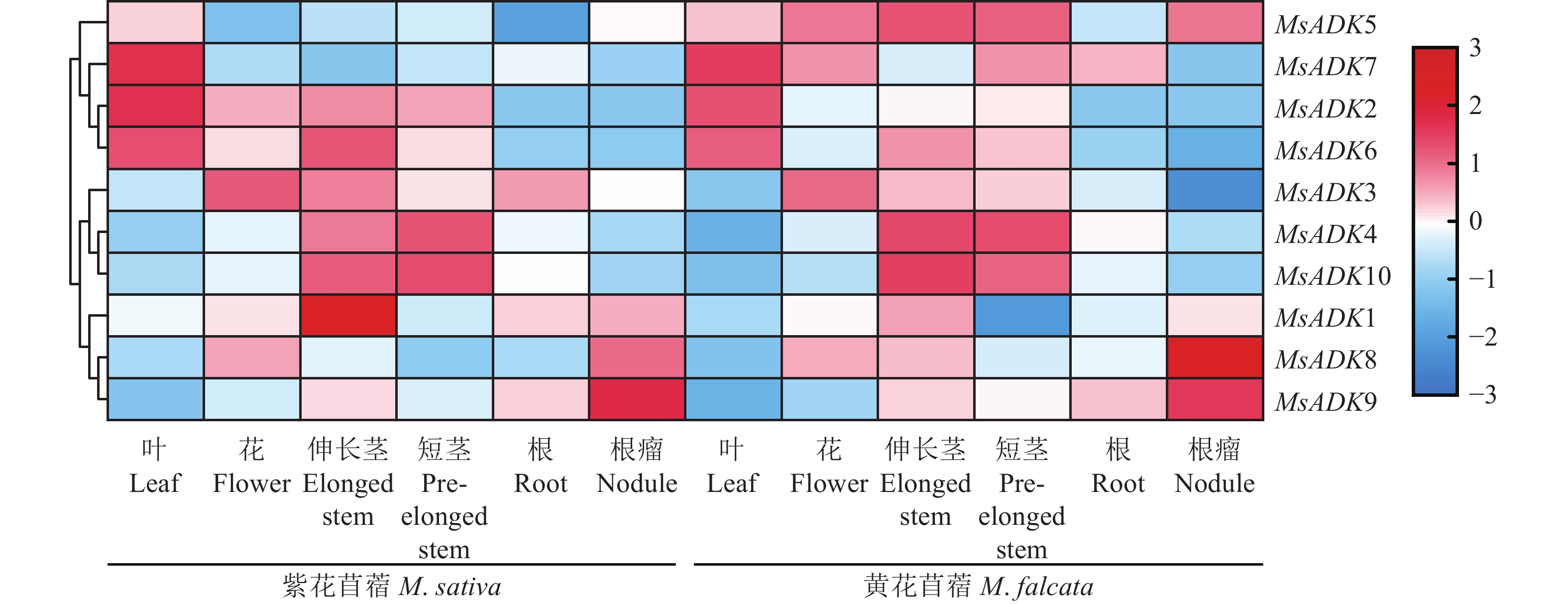
分析了MsADK基因在紫花苜蓿和黄花苜蓿不同组织中的表达量,MsADK基因在两个品种间具有相同的表达模式(图7)。MsADK1、MsADK3、MsADK7基因在不同组织间的表达量一致。MsADK5、MsADK8、MsADK9基因在所有组织中均有较高的表达水平,尤其在根瘤中高表达。MsADK2的表达具有组织特异性,在根瘤和根中均不表达,而在叶片中的表达量最高。同样,MsADK6在地上组织中的表达量要显著的高于地下组织。MsADK4和MsADK10则在茎中的表达量高于其他组织。表明MsADK基因在调节紫花苜蓿生长发育中发挥不同的作用。
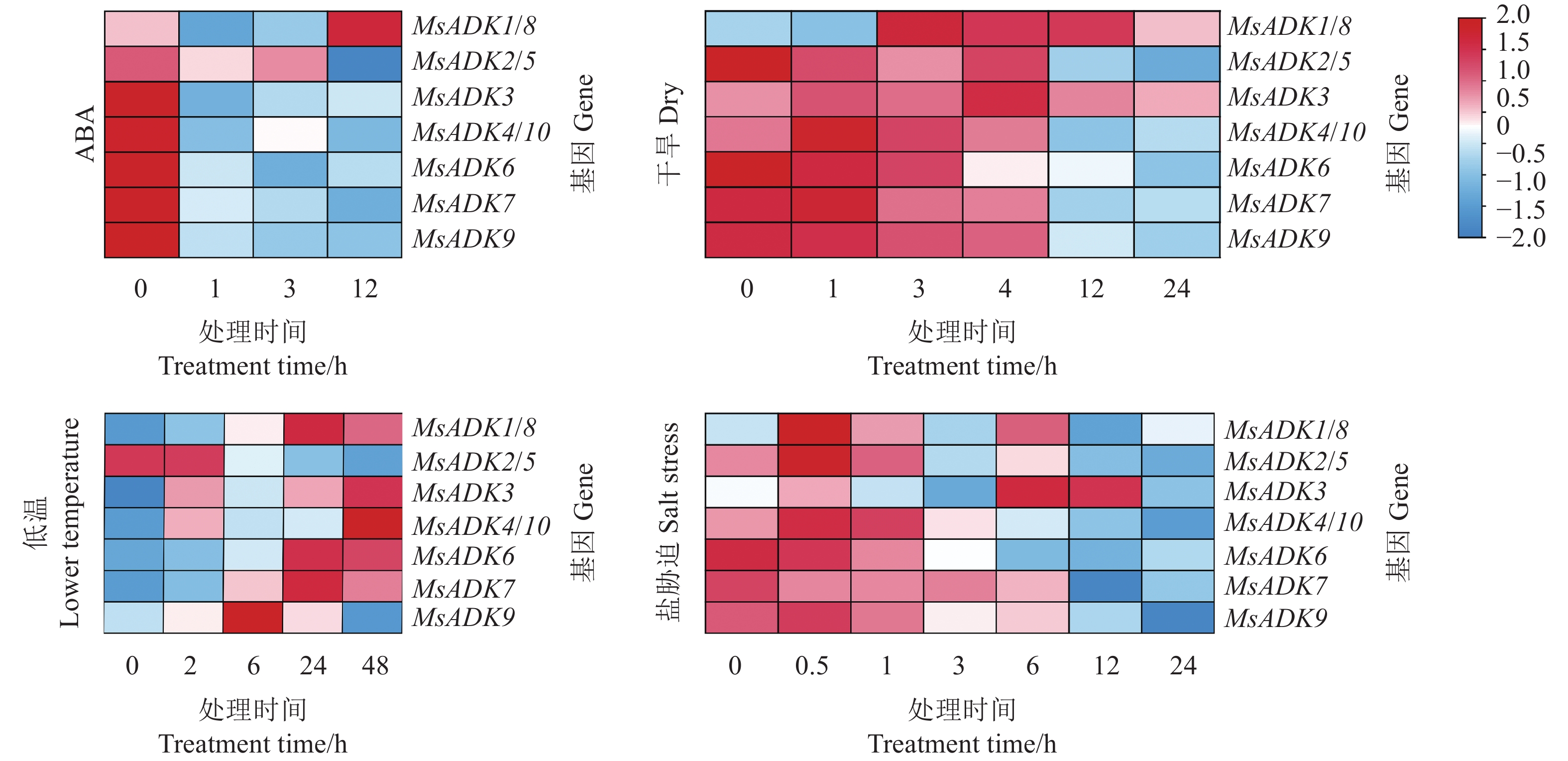
为了进一步解析MsADK基因在不同非生物胁迫的潜在功能,分析了它们分别在ABA、干旱、低温和盐胁迫下的转录表达谱(图8)。用ABA处理紫花苜蓿3 h后,除MsADK1和MsADK8在12 h表达量增加,剩余MsADKs基因的表达量在各处理时间段均明显下调。同样,在干旱和盐胁迫下,MsADK4、MsADK6、MsADK7、MsADK9和MADKs10的表达在处理早期(干旱胁迫:1~6 h;盐胁迫:0.5~1 h)和对照保持相对一致的表达量,但随着处理时间的延长,上述5个基因明显下调。在两种渗透胁迫下,MsADK1、MsADK3和MsADK8随着处理时间的增加,均出现了不同程度的上调表达,其中MsADK1和MsADK8对胁迫的反应更为敏感。值得注意的是,低温胁迫下MsADK基因表现出了不同于其他非生物胁迫的表达模式,除了MsADK2、MsADK5下调表达外,其余基因从6 h开始,出现不同程度的上调表达。
3. 讨论
腺苷酸类化合物在生物体内的含量变化被认为是引起细胞能量代谢的主要因素,其中腺苷酸激酶是生成ADP和平衡磷酸化反应的关键酶,在细胞质、线粒体和叶绿体等细胞器中均有分布[20, 27]。研究表明ADK基因的转录表达,将直接影响腺苷代谢库容大小。目前已从大豆、甘薯(Dioscorea esculenta)、番茄等多种植物种中克隆了ADK基因。然而,尚未在紫花苜蓿中对ADK基因家族进行全基因组水平的鉴定与分析。在紫花苜蓿、拟南芥、水稻、蒺藜苜蓿、番茄和马铃薯中分别鉴定到10、7、10、7、11和12个ADK基因,不同物种间ADK基因的数目接近,表明ADK基因在物种间高度保守,与物种基因组大小无关。用拟南芥、水稻、蒺藜苜蓿和紫花苜蓿的ADK蛋白构建系统进化树,将34个ADK蛋白划分为5个亚家族,每个亚家族均包含了上述4个物种的ADK蛋白。蒺藜苜蓿和紫花苜蓿间表现出了高度的同源性,且每个紫花苜蓿的ADK蛋白都有一个高度同源的蒺藜苜蓿蛋白。此外,单双子叶植物间无明显的分离,表明ADK蛋白在单双子叶植物分化之前就已存在。紫花苜蓿MsADK基因的亚细胞定位多样,在叶绿体、线粒体、细胞质、细胞核等细胞器中均有分布,主要位于细胞质中(40%),与番茄和木薯(Manihot esculenta)中主要定位于线粒体中的研究结果不同[14, 20]。基序分析显示Motif 1和Motif 3在所有MsADK蛋白序列中都含有,但是不同亚家族成员间含有特异的基序。同样,聚类到相同亚家族成员的外显子数目接近,推测MsADK成员间可能存在功能差异。
非生物胁迫是影响全球农作物产量的主要原因,每年约有50%的作物减产是由非生物胁迫造成的。ADK除了直接影响能量代谢外,还影响植物的其他方面,包括生长、发育、开花、抗逆等[11, 27]。研究表明ADK基因的下调将会导致腺苷酸库积累增加,进而影响亚细胞的氧化还原状态,并刺激AGPase (淀粉合成的关键酶)的二硫键断裂,将其从非活性形式转化为活性形式[28-29]。拟南芥细胞核定位的AAK6基因调控茎的生长速度[11];马铃薯质体中的ADK基因下调表达,使淀粉的积累量增加60%[14]。紫花苜蓿MsADK基因在不同组织中均有表达,茎中的表达量普遍高于其他组织,其中MsADK5、MsADK8和MsADK9在根瘤中高度表达,表明MsADK基因在调控紫花苜蓿地上和地下组织的生长发育中均发挥重要功能。
通过分析ADK在干旱、盐、ABA和低温胁迫下的表达模式,MsADK1和MsADK8在所有非生物胁迫下具有明显的上调表达外,多数MsADK基因在干旱、盐和ABA胁迫下与低温胁迫呈现相反的表达模式,如MsADK4,MsADK7,MsADK9和MsADK10等在干旱、盐和ABA胁迫中下调表达,但是在低温胁迫下上调表达。MsADK基因与大豆间的共线性关系最强,有研究分析了耐盐(南农1138-2)和盐敏感(科丰1号)品种在盐胁迫下GmADK基因的表达模式,结果表明GmADK基因在盐敏感品种中表达量降低,而在耐盐品种中升高[17]。在番茄的研究中表明,大多数SlADK基因受干旱诱导上调表达[20]。非生物胁迫下,ADK基因在不同物种间的表达模式存在差异,具有正向和负向两种调控机制,推测可能与选择的研究材料和胁迫类型有关,精确的调控机理还有待进一步深入研究。
4. 结论
本研究在全基因组水平鉴定了紫花苜蓿ADK基因家族,并对其理化性质、基因结构、进化关系、顺式作用元件等进行分析。在此基础上,对其不同组织和不同非生物胁迫下的表达模式进行分析,在不同胁迫类型下ADK基因表现出正、负两种调控机制。MsADK基因在进化过程中高度保守性,参与调控紫花苜蓿生长发育和逆境胁迫响应,本研究对于MsADK成员功能验证,将为紫花苜蓿基于基因工程的遗传改良提供一定基础。
参考文献
[1] REGIERER B, FERNIE A R, SPRINGER F, PEREZ-MELIS A, LEISSE A, KOEHL K, WILLMITZER L, GEIGENBERGER P, KOSSMANN J. Starch content and yield increase as a result of altering adenylate pools in transgenic plants. Nature Biotechnology, 2002, 20(12): 1256-1260. doi: 10.1038/nbt760
[2] LANGE P R, GESERICK C, TISCHENDORF G, ZRENNER R. Functions of chloroplastic adenylate kinases in Arabidopsis. Plant Physiology, 2007, 146(2): 323-324.
[3] CHING T M, CRANE J M, STAMP D L. Adenylate energy pool and energy charge in maturing rape seeds. Plant Physiology, 1974, 54(5): 748-751. doi: 10.1104/pp.54.5.748
[4] RAYMOND P, PRADET A. Stabilization of adenine nucleotide ratios at various values by an oxygen limitation of respiration in germinating lettuce (Lactuca sativa) seeds. Biochemical Journal, 1980, 190(1): 39-44. doi: 10.1042/bj1900039
[5] 叶纯. 分子动力学模拟研究腺苷酸激酶催化循环及PLA1脂肪酶底物识别机制. 合肥: 中国科学技术大学博士学位论文, 2019. YE C. Molecular dynamics simulation studies on the adenylate kinase catalytic cycle and the substrate recognition mechanism of lipase PLA1. PhD Thesis. Hefei: University of Science and Technology of China, 2019.
[6] BIRKENHEAD K, WALKER D, FOYER C. The intracellular distribution of adenylate kinase in the leaves of spinach, wheat and barley. Planta, 1982, 156(2): 171-175. doi: 10.1007/BF00395432
[7] THIEULIN-PARDO G, SCHRAMM A, LIGNON S, LEBRUN R, KOJADINOVIC M, GONTERO B. The intriguing CP12-like tail of adenylate kinase 3 from chlamydomonas reinhardtii. FEBS Journal, 2016, 283(18): 3389-3407. doi: 10.1111/febs.13814
[8] 曾令江, 陈敏, 潘夕春, 张启堂, 廖志华. 马铃薯腺苷酸激酶基因的克隆分析及其植物表达载体的构建. 生物技术通讯, 2007, 18(3): 405-408. doi: 10.3969/j.issn.1009-0002.2007.03.013 ZENG L J, CHEN M, PAN X C, ZHANG Q T, LIAO Z H. Cloning, characterization and constructing of plant expression vector of adenylate kinase gene from potato. Letters in Biotechnology, 2007, 18(3): 405-408. doi: 10.3969/j.issn.1009-0002.2007.03.013
[9] HAMPP R D, GOLLER M, ZIEGLER H. Adenylate levels, energy charge, and phosphorylation potential during dark-light and light-dark transition in chloroplasts, mitochondria, and cytosol of mesophyll protoplasts from Avena sativa L. Plant Physiology, 1982, 69(2): 448-455. doi: 10.1104/pp.69.2.448
[10] KAWAI M, UCHIMIYA H. Biochemical properties of rice adenylate kinase and subcellular location in plant cells. Plant Molecular Biology, 1995, 27(5): 943-951. doi: 10.1007/BF00037022
[11] FENG X, YANG R, ZHENG X, ZHANG F. Identification of a novel nuclear-localized adenylate kinase 6 from Arabidopsis thaliana as an essential stem growth factor. Plant Physiology and Biochemistry, 2012, 61: 180-186. doi: 10.1016/j.plaphy.2012.10.002
[12] CARRARI F, COLL-GARCIA D, SCHAUER N, LYTOVCHENKO A, PALACIOS-ROJAS N, BALBO I, ROSSO M, FERNIE A R. Deficiency of a plastidial adenylate kinase in Arabidopsis results in elevated photosynthetic amino acid biosynthesis and enhanced growth. Plant Physiology, 2005, 137(1): 70-82. doi: 10.1104/pp.104.056143
[13] LANGE PR, GESERICK C, TISCHENDORF G, ZRENNER R. Functions of chloroplastic adenylate kinases in Arabidopsis. Plant Physiology, 2008, 146(2): 492-504.
[14] BOONRUENG C, TANGPRANOMKORN S, YAZHISAI U, SIRIKANTARAMAS S. Molecular cloning, subcellular localization and characterization of two adenylate kinases from cassava, Manihot esculenta Crantz cv. KU50. Journal of Plant Physiology, 2016, 204: 66-73. doi: 10.1016/j.jplph.2016.08.001
[15] ZHANG Y, LAUNAY H, LIU F, LEBRUN R, GONTERO B. Interaction between adenylate kinase 3 and glyceraldehyde-3-phosphate dehydrogenase from Chlamydomonas reinhardtii. FEBS Journal, 2018, 285(13): 2495-2503. doi: 10.1111/febs.14494
[16] RAVENEAU M-P, BENAMAR A, MACHEREL D. Water content, adenylate kinase, and mitochondria drive adenylate balance in dehydrating and imbibing seeds. Journal of Experimental Botany, 2017, 68(13): 3501-3512. doi: 10.1093/jxb/erx182
[17] 盖江涛, 赵团结, 李艳, 盖钧镒. 大豆腺苷酸激酶基因GmADK的克隆与表达分析. 作物学报, 2013, 39(10): 1739-1745. doi: 10.3724/SP.J.1006.2013.01739 GAI J T, ZHAO T J, LI Y, GAI J Y. Cloning and expression analysis of an adenylate kinase gene GmADK in soybean. Acta Agronomica Sinica, 2013, 39(10): 1739-1745. doi: 10.3724/SP.J.1006.2013.01739
[18] GONG P, ZHANG J, LI H, YANG C, ZHANG C, ZHANG X, KHURRAM Z, ZHANG Y, WANG T, FEI Z, YE Z. Transcriptional profiles of drought-responsive genes in modulating transcription signal transduction, and biochemical pathways in tomato. Journal of Experimental Botany, 2010, 61(13): 3563-3575. doi: 10.1093/jxb/erq167
[19] LANGE P R, GESERICK C, TISCHENDORF G, ZRENNER R. Functions of chloroplastic adenylate ases in Arabidopsis, 2008, 146, 492-504.
[20] YANG L, CAO H, ZHANG X, GUI L, CHEN Q, QIAN G, XIAO J, LI Z. Genome-wide identification and expression analysis of tomato ADK gene family during development and stress. International Journal of Molecular Sciences, 2021, 22(14): 7708. doi: 10.3390/ijms22147708
[21] 闵学阳, 刘文献, 张正社, 韦兴燚, 齐晓, 张义, 刘志鹏, 王彦荣. 苜蓿DUS测试标准品种SSR分子标记指纹图谱的构建. 草业学报, 2017, 26(11): 47-56. doi: 10.11686/cyxb2017038 MIN X Y, LIU W X, ZHANG Z S, WEI X Y, QI X, ZHANG Y, LIU Z P, WANG Y R. Construction of SSR marker fingerprint database of standard alfalfa varieties utilizing DUS tests. Prataculturae Sinica, 2017, 26(11): 47-56. doi: 10.11686/cyxb2017038
[22] CHEN C, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. doi: 10.1016/j.molp.2020.06.009
[23] O’ROURKE J A, FU F, BUCCIARELLI B, YANG S S, SAMAC D A, LAMB J F S, MONTEROS M J, GRAHAM M A, GRONWALD J W, KROM N, LI J, DAI X, ZHAO P X, VANCE C P. The Medicago sativa gene index 1.2: A web-accessible gene expression atlas for investigating expression differences between Medicago sativa subspecies. BMC Genomics, 2015, 16(1): 502. doi: 10.1186/s12864-015-1718-7
[24] LUO D, ZHOU Q, WU Y, CHAI X, LIU W, WANG Y, YANG Q, WANG Z, LIU Z. Full-length transcript sequencing and comparative transcriptomic analysis to evaluate the contribution of osmotic and ionic stress components towards salinity tolerance in the roots of cultivated alfalfa (Medicago sativa L.). BMC Plant Biology, 2019, 19(1): 32. doi: 10.1186/s12870-019-1630-4
[25] LUO D, WU Y, LIU J, ZHOU Q, LIU W, WANG Y, YANG Q, WANG Z, LIU Z. Comparative transcriptomic and physiological analyses of Medicago sativa L. indicates that multiple regulatory networks are activated during continuous ABA treatment. International Journal of Molecular Sciences, 2019, 20(1): 47.
[26] ZHOU Q, LUO D, CHAI X, WU Y, WANG Y, NAN Z, YANG Q, LIU W, LIU Z. Multiple regulatory networks are activated during cold stress in Medicago sativa L. International Journal of Molecular Sciences, 2018, 19(10): 3169. doi: 10.3390/ijms19103169
[27] IGAMBERDIEV A U, KLECZKOWSKI L A. Equilibration of adenylates in the mitochondrial intermembrane space maintains respiration and regulates cytosolic metabolism. Journal of Experimental Botany, 2006, 57(10): 2133-2141. doi: 10.1093/jxb/erl006
[28] TIESSEN A, HENDRIKS J H M, STITT M, BRANSCHEID A, GIBON Y, FARRÉ E M, GEIGENBERGER P. Starch synthesis in potato tubers is regulated by post-translational redox modification of ADP-glucose pyrophosphorylase: A novel regulatory mechanism linking starch synthesis to the sucrose supply. The Plant Cell, 2002, 14(9): 2191-2213. doi: 10.1105/tpc.003640
[29] BALLICORA M A, FRUEAUF J B, FU Y, SCHÜRMANN P, PREISS J. Activation of the potato tuber ADP-glucose pyrophosphorylase by thioredoxin. Journal of Biological Chemistry, 2000, 275(2): 1315-1320. doi: 10.1074/jbc.275.2.1315
-
图 1 紫花苜蓿、蒺藜苜蓿、拟南芥和水稻ADK蛋白的系统进化分析
图中正方形、三角形、圆形和菱形分别代表紫花苜蓿、水稻、蒺藜苜蓿和拟南芥。
Figure 1. Phylogenetic tree of ADK genes from Medicago sativa, M. truncatula, Arabidopsis thaliana, and Oryza sativa
Squares, triangles, circles and diamonds represent Medicago sativa, Oryza sativa, M. truncatula, and, Arabidopsis thaliana, respectively.
表 1 紫花苜蓿ADK基因家族信息
Table 1 Information of ADK gene family in Medicago sativa
基因
名称
Gene
Name基因号
Gene
ID染色体
Chr.
No蛋白
长度
Amino acids
length/aacDNA
长度
cDNA
length/bp蛋白质
分子量
Protein molecular
weight/kDa等电
点
pI疏水性
指数
Protein
GRAVY亚细胞
定位
Subcellular
localization蛋白稳定
指数
Instability
index拟南芥
同源基因
Homologous gene
of ArabidopsisMsADK1 MS.gene029884.t1 chr1.4 280 843 30.75 6.67 −0.164 叶绿体
Chloroplast44.68 AT2G37250.1 MsADK2 MS.gene81523.t1 chr2.2 504 1515 56.79 6.41 −0.382 细胞质
Cytoplasmic42.45 AT5G35170.1 MsADK3 MS.gene045674.t1 chr4.3 244 735 27.71 8.64 −0.312 叶绿体
Chloroplast49.23 AT4G25280.2 MsADK4 MS.gene77729.t1 chr4.4 242 729 27.52 7.29 −0.324 过氧化体
Peroxisomal52.32 AT4G25280.2 MsADK5 MS.gene97107.t1 chr4.4 234 705 26.11 6.53 −0.311 细胞质
Cytoplasmic35.63 AT5G47840.1 MsADK6 MS.gene060445.t1 chr7.1 265 798 29.90 6.80 −0.448 细胞核
Nuclear50.29 AT3G01820.1 MsADK7 MS.gene007478.t1 chr7.1 268 807 219.28 7.39 −0.206 线粒体
Mitochondrial53.86 AT2G37250.1 MsADK8 MS.gene09796.t1 chr7.3 246 738 27.04 9.33 −0.150 线粒体
Mitochondrial48.54 AT2G37250.1 MsADK9 MS.gene011389.t1 chr8.3 243 732 26.67 7.97 −0.326 细胞质
Cytoplasmic35.91 AT5G63400.1 MsADK10 MS.gene39664.t1 chr8.3 208 627 23.15 5.46 −0.466 细胞质
Cytoplasmic43.44 AT5G26667.4 pI:等电点; GRAVY:疏水性指数。
pI: isoelectric; GRAVY: grand average of hydropathicity.表 2 MsADK蛋白二级结构
Table 2 Secondary structure of MsADK proteins
基因名称
Gene nameα-螺旋 α-helix β-转角 β-turn 无规则卷曲 Random coil 延伸链 Extended chain 数量
Number占比
Ratio/%数量
Number占比
Ratio/%数量
Number占比
Ratio/%数量
Number占比
Ratio/%MsADK1 125 44.64 27 9.64 98 35.00 30 10.71 MsADK2 213 46.26 41 8.13 180 35.71 70 13.89 MsADK3 113 46.31 16 6.56 84 34.43 31 12.70 MsADK4 130 53.72 18 7.44 67 27.69 27 11.16 MsADK5 121 51.71 21 8.97 64 27.35 28 11.97 MsADK6 134 50.57 21 7.92 79 29.81 31 11.70 MsADK7 111 41.42 23 8.58 105 39.18 29 10.82 MsADK8 92 37.40 14 5.69 113 45.93 27 10.98 MsADK9 111 45.68 25 10.29 79 32.51 28 11.52 MsADK10 97 46.63 19 9.13 65 31.25 27 12.98 平均值 Mean 124.7 46.43 22.5 8.24 93.4 33.89 32.8 11.84 -
[1] REGIERER B, FERNIE A R, SPRINGER F, PEREZ-MELIS A, LEISSE A, KOEHL K, WILLMITZER L, GEIGENBERGER P, KOSSMANN J. Starch content and yield increase as a result of altering adenylate pools in transgenic plants. Nature Biotechnology, 2002, 20(12): 1256-1260. doi: 10.1038/nbt760
[2] LANGE P R, GESERICK C, TISCHENDORF G, ZRENNER R. Functions of chloroplastic adenylate kinases in Arabidopsis. Plant Physiology, 2007, 146(2): 323-324.
[3] CHING T M, CRANE J M, STAMP D L. Adenylate energy pool and energy charge in maturing rape seeds. Plant Physiology, 1974, 54(5): 748-751. doi: 10.1104/pp.54.5.748
[4] RAYMOND P, PRADET A. Stabilization of adenine nucleotide ratios at various values by an oxygen limitation of respiration in germinating lettuce (Lactuca sativa) seeds. Biochemical Journal, 1980, 190(1): 39-44. doi: 10.1042/bj1900039
[5] 叶纯. 分子动力学模拟研究腺苷酸激酶催化循环及PLA1脂肪酶底物识别机制. 合肥: 中国科学技术大学博士学位论文, 2019. YE C. Molecular dynamics simulation studies on the adenylate kinase catalytic cycle and the substrate recognition mechanism of lipase PLA1. PhD Thesis. Hefei: University of Science and Technology of China, 2019.
[6] BIRKENHEAD K, WALKER D, FOYER C. The intracellular distribution of adenylate kinase in the leaves of spinach, wheat and barley. Planta, 1982, 156(2): 171-175. doi: 10.1007/BF00395432
[7] THIEULIN-PARDO G, SCHRAMM A, LIGNON S, LEBRUN R, KOJADINOVIC M, GONTERO B. The intriguing CP12-like tail of adenylate kinase 3 from chlamydomonas reinhardtii. FEBS Journal, 2016, 283(18): 3389-3407. doi: 10.1111/febs.13814
[8] 曾令江, 陈敏, 潘夕春, 张启堂, 廖志华. 马铃薯腺苷酸激酶基因的克隆分析及其植物表达载体的构建. 生物技术通讯, 2007, 18(3): 405-408. doi: 10.3969/j.issn.1009-0002.2007.03.013 ZENG L J, CHEN M, PAN X C, ZHANG Q T, LIAO Z H. Cloning, characterization and constructing of plant expression vector of adenylate kinase gene from potato. Letters in Biotechnology, 2007, 18(3): 405-408. doi: 10.3969/j.issn.1009-0002.2007.03.013
[9] HAMPP R D, GOLLER M, ZIEGLER H. Adenylate levels, energy charge, and phosphorylation potential during dark-light and light-dark transition in chloroplasts, mitochondria, and cytosol of mesophyll protoplasts from Avena sativa L. Plant Physiology, 1982, 69(2): 448-455. doi: 10.1104/pp.69.2.448
[10] KAWAI M, UCHIMIYA H. Biochemical properties of rice adenylate kinase and subcellular location in plant cells. Plant Molecular Biology, 1995, 27(5): 943-951. doi: 10.1007/BF00037022
[11] FENG X, YANG R, ZHENG X, ZHANG F. Identification of a novel nuclear-localized adenylate kinase 6 from Arabidopsis thaliana as an essential stem growth factor. Plant Physiology and Biochemistry, 2012, 61: 180-186. doi: 10.1016/j.plaphy.2012.10.002
[12] CARRARI F, COLL-GARCIA D, SCHAUER N, LYTOVCHENKO A, PALACIOS-ROJAS N, BALBO I, ROSSO M, FERNIE A R. Deficiency of a plastidial adenylate kinase in Arabidopsis results in elevated photosynthetic amino acid biosynthesis and enhanced growth. Plant Physiology, 2005, 137(1): 70-82. doi: 10.1104/pp.104.056143
[13] LANGE PR, GESERICK C, TISCHENDORF G, ZRENNER R. Functions of chloroplastic adenylate kinases in Arabidopsis. Plant Physiology, 2008, 146(2): 492-504.
[14] BOONRUENG C, TANGPRANOMKORN S, YAZHISAI U, SIRIKANTARAMAS S. Molecular cloning, subcellular localization and characterization of two adenylate kinases from cassava, Manihot esculenta Crantz cv. KU50. Journal of Plant Physiology, 2016, 204: 66-73. doi: 10.1016/j.jplph.2016.08.001
[15] ZHANG Y, LAUNAY H, LIU F, LEBRUN R, GONTERO B. Interaction between adenylate kinase 3 and glyceraldehyde-3-phosphate dehydrogenase from Chlamydomonas reinhardtii. FEBS Journal, 2018, 285(13): 2495-2503. doi: 10.1111/febs.14494
[16] RAVENEAU M-P, BENAMAR A, MACHEREL D. Water content, adenylate kinase, and mitochondria drive adenylate balance in dehydrating and imbibing seeds. Journal of Experimental Botany, 2017, 68(13): 3501-3512. doi: 10.1093/jxb/erx182
[17] 盖江涛, 赵团结, 李艳, 盖钧镒. 大豆腺苷酸激酶基因GmADK的克隆与表达分析. 作物学报, 2013, 39(10): 1739-1745. doi: 10.3724/SP.J.1006.2013.01739 GAI J T, ZHAO T J, LI Y, GAI J Y. Cloning and expression analysis of an adenylate kinase gene GmADK in soybean. Acta Agronomica Sinica, 2013, 39(10): 1739-1745. doi: 10.3724/SP.J.1006.2013.01739
[18] GONG P, ZHANG J, LI H, YANG C, ZHANG C, ZHANG X, KHURRAM Z, ZHANG Y, WANG T, FEI Z, YE Z. Transcriptional profiles of drought-responsive genes in modulating transcription signal transduction, and biochemical pathways in tomato. Journal of Experimental Botany, 2010, 61(13): 3563-3575. doi: 10.1093/jxb/erq167
[19] LANGE P R, GESERICK C, TISCHENDORF G, ZRENNER R. Functions of chloroplastic adenylate ases in Arabidopsis, 2008, 146, 492-504.
[20] YANG L, CAO H, ZHANG X, GUI L, CHEN Q, QIAN G, XIAO J, LI Z. Genome-wide identification and expression analysis of tomato ADK gene family during development and stress. International Journal of Molecular Sciences, 2021, 22(14): 7708. doi: 10.3390/ijms22147708
[21] 闵学阳, 刘文献, 张正社, 韦兴燚, 齐晓, 张义, 刘志鹏, 王彦荣. 苜蓿DUS测试标准品种SSR分子标记指纹图谱的构建. 草业学报, 2017, 26(11): 47-56. doi: 10.11686/cyxb2017038 MIN X Y, LIU W X, ZHANG Z S, WEI X Y, QI X, ZHANG Y, LIU Z P, WANG Y R. Construction of SSR marker fingerprint database of standard alfalfa varieties utilizing DUS tests. Prataculturae Sinica, 2017, 26(11): 47-56. doi: 10.11686/cyxb2017038
[22] CHEN C, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. doi: 10.1016/j.molp.2020.06.009
[23] O’ROURKE J A, FU F, BUCCIARELLI B, YANG S S, SAMAC D A, LAMB J F S, MONTEROS M J, GRAHAM M A, GRONWALD J W, KROM N, LI J, DAI X, ZHAO P X, VANCE C P. The Medicago sativa gene index 1.2: A web-accessible gene expression atlas for investigating expression differences between Medicago sativa subspecies. BMC Genomics, 2015, 16(1): 502. doi: 10.1186/s12864-015-1718-7
[24] LUO D, ZHOU Q, WU Y, CHAI X, LIU W, WANG Y, YANG Q, WANG Z, LIU Z. Full-length transcript sequencing and comparative transcriptomic analysis to evaluate the contribution of osmotic and ionic stress components towards salinity tolerance in the roots of cultivated alfalfa (Medicago sativa L.). BMC Plant Biology, 2019, 19(1): 32. doi: 10.1186/s12870-019-1630-4
[25] LUO D, WU Y, LIU J, ZHOU Q, LIU W, WANG Y, YANG Q, WANG Z, LIU Z. Comparative transcriptomic and physiological analyses of Medicago sativa L. indicates that multiple regulatory networks are activated during continuous ABA treatment. International Journal of Molecular Sciences, 2019, 20(1): 47.
[26] ZHOU Q, LUO D, CHAI X, WU Y, WANG Y, NAN Z, YANG Q, LIU W, LIU Z. Multiple regulatory networks are activated during cold stress in Medicago sativa L. International Journal of Molecular Sciences, 2018, 19(10): 3169. doi: 10.3390/ijms19103169
[27] IGAMBERDIEV A U, KLECZKOWSKI L A. Equilibration of adenylates in the mitochondrial intermembrane space maintains respiration and regulates cytosolic metabolism. Journal of Experimental Botany, 2006, 57(10): 2133-2141. doi: 10.1093/jxb/erl006
[28] TIESSEN A, HENDRIKS J H M, STITT M, BRANSCHEID A, GIBON Y, FARRÉ E M, GEIGENBERGER P. Starch synthesis in potato tubers is regulated by post-translational redox modification of ADP-glucose pyrophosphorylase: A novel regulatory mechanism linking starch synthesis to the sucrose supply. The Plant Cell, 2002, 14(9): 2191-2213. doi: 10.1105/tpc.003640
[29] BALLICORA M A, FRUEAUF J B, FU Y, SCHÜRMANN P, PREISS J. Activation of the potato tuber ADP-glucose pyrophosphorylase by thioredoxin. Journal of Biological Chemistry, 2000, 275(2): 1315-1320. doi: 10.1074/jbc.275.2.1315
-
期刊类型引用(2)
1. 韩硕,韩晓文,胡义锋,杨静美,陈中义,朱永兴,尹军良. 入侵杂草喜旱莲子草WRKY转录因子家族鉴定及其除草剂胁迫表达特征. 草业科学. 2024(11): 2517-2534 .  本站查看
本站查看
2. 韩晓文,韩硕,胡义锋,王梦如,陈中义,朱永兴,尹军良. 空心莲子草ApCAT基因家族鉴定和表达模式. 草业科学. 2023(11): 2889-2901 .  本站查看
本站查看
其他类型引用(2)



 下载:
下载: